Autophagie
Macroautophagie
Formation de l'autophagosome
Nucléation du phagophore
- Vue d'ensemble du système endomembranaire
- Autophagie
- Système UPS (Système UPS (Ubiquitine-protéasome))
- Fusion et fission membranaire
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

La nucléation du phagophore nécessite le recrutement et l’activation du complexe PI3KC3-CI, activé par le complexe ULK/Atg1, responsable de la production de PI(3)P sur les omégasomes.
La biogenèse des autophagosomes implique plusieurs processus :
- l'initiation du phagophore au niveau des omégasomes,
- sa nucléation,
- son élongation,
- sa fermeture pour former un autophagosome.

Vous pouvez lire : Autophagosome biogenesis: From membrane growth to closure (2020), Emerging roles of ATG proteins and membrane lipids in autophagosome formation (2020) et Structural view on autophagosome formation (2023), ce dernier article analyse les structures des différents composants.
Nucléation du phagophore
Activation du complexe PI3KC3-CI
Une fois le complexe ULK recruté et activé au niveau de l’omégasome, celui-ci permet le recrutement et l’activation du complexe PI3KC3-CI (Vacuolar protein sorting 34/phosphatidylinositol 3-kinase de classe 3), responsable de la production de PI(3)P, nécessaire à la nucléation du phagophore, i.e. à son développement.

(Figure : vetopsy.fr d'après Nishimura et Tooze)
1. Le complexe PI3KC3-CI comprend plusieurs protéines :
- VPS34/Vps34,
- VPS15/p150/Vps15,
- Beclin1(BECN1)//Vps30,
- ATG14L/Atg14,
- Atg38/NRBF2, non représentée sur la figure ci-dessus, i.e. cinquième sous-unité qui facilite la formation du complexe PI3KC3-C1 et induit en outre sa dimérisation.

Le complexe PI3KC3-CI et ses composants sont étudiés dans des chapitres spéciaux.
2. ULK phosphoryle Beclin1 et Vps34 pour le recrutement du complexe (A molecular perspective of mammalian autophagosome biogenesis 2018).

(Figure : vetopsy.fr d'après Melia et coll)
a. La stabilisation de la localisation membranaire du PIK3C3-CI est assurée par l'interaction directe entre ATG13 du complexe ULK et ATG14 (The ULK1 complex mediates MTORC1 signaling to the autophagy initiation machinery via binding and phosphorylating ATG14 2016).
b. Le ciblage de PIK3C3-CI vers la membrane du RE est facilité par plusieurs sous-unités qui forment deux bras :
- un domaine BARA (Beta-Alpha-Repeated, Autophagy specific) dans BECN1,
- un domaine riche en cystéine N-terminal et un domaine BATS dans ATG14,
- le domaine C-terminal, domaine hélicoïdal et lipide-kinase de Vps34,
- le domaine N-terminal de myristoylation de VPS15.
c. L'extrémité C-terminale de VPS34 détermine l'orientation du complexe PI3KC3-C1, tandis que le domaine BATS d'ATG14L1 est essentiel pour détecter la courbure de la membrane et médier la spécificité des lipides aux membranes cibles (Cryo-EM structure and biochemical analysis reveal the basis of the functional difference between human PI3KC3-C1 and -C2 2017).

(Figure : vetopsy.fr d'après Melia et coll)
2. D’autres protéines peuvent, selon les modèles, stimuler l’activation de ce complexe.
a. AMBRA1 (Activating Molecule in Beclin 1-Regulated Autophagy), liée à TRFF6 une ubiquitinase, par l'ubiquitinilation de ULK1 au niveau de sa lysine 63, promeut son interaction avec Vps34, mais aussi provoque la libération de BECN1/Vps34 du cytosquelette et leur recrutement au RE (Ambra1 at a glance 2015).
b. UVRAG/Vps38 (UV-radiation resistance-associated gene) favorise l’activation du complexe de nucléation, la formation de l’autophagosome et sa maturation.
- UVRAG est impliqué dans l’initiation de la réticulophagie, indépendamment de sa fonction canonique dans le complexe PI3KC3, en interagissant avec des récepteurs du réticulum endoplasmique (RE) comme FAM134B, ATL3 et RTN3L.
- Il favorise la dégradation des feuillets de RE, des tubules et des protéines luminales du RE sujettes à l’agrégation, telles que la proinsuline pathogène (UVRAG cooperates with cargo receptors to assemble the ER‐phagy site 2023).
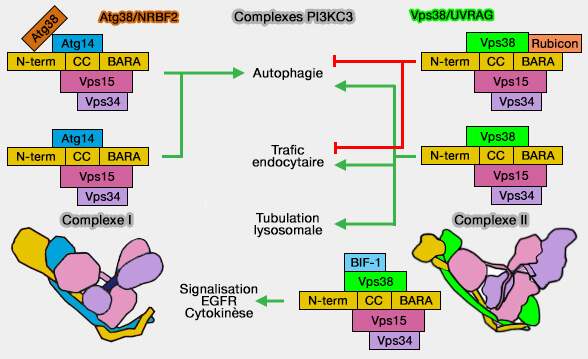
(Figure : vetopsy.fr d'après Tran et coll)
3. A l’inverse, Rubicon (RUN domain and cysteine-rich domain containing, Beclin1-interacting protein), inhibe l’activité du complexe Vps34/Beclin-1 et de ce fait la nucléation du phagophore (Structural basis for autophagy inhibition by the human Rubicon–Rab7 complex 2020).
Rubicon stabiliserait la boucle β1 de BECN1 et bloquerait l’amarrage membranaire (Bidirectional Control of Autophagy by BECN1 BARA Domain Dynamics 2019).
Remarque : les composants du complexe PI3KC3-C1, VPS34, ATG14 et BECLIN1 contiennent également des motifs LIR fonctionnels et interagissent préférentiellement avec GABARAP et GABARAPL1 (Members of the autophagy class III phosphatidylinositol 3-kinase complex I interact with GABARAP and GABARAPL1 via LIR motifs 2019).
Comme le motif LIR dans l'ATG14L est en position étroite à son domaine BATS, ces deux motifs pourraient fonctionner comme un détecteur de coïncidence pour le ciblage spécifique des membranes autophagiques.
Fonctions de PI(3)P dans la nucléation
Le RE contient très peu de PI(3)P dans des conditions basales et sa production locale recrute des protéines spécifiques au niveau du phagophore (Lipids, lysosomes, and autophagy 2016).

Vous pouvez lire : Lipids and membrane-associated proteins in autophagy (2020), qui comprend un tableau récapitulant toutes les protéines autophagiques qui se lient directement à la membrane pendant toutes les phases de la formation de l'autophagosome.
Protéines Atg18/WIPI
1. PI(3)P au niveau de l'omegasome permet le recrutement de membres de la famille de protéines Atg18/WIPI1-4, fonctionnellement liées à Atg18 de la levure par :
- deux poches de liaison au PI(3)P au niveau des lames 5 et 6,
- une hélice dans la lame 6 qui sert d'ancrage membranaire s'insérant profondément dans la bicouche lipidique.

(Figure : vetopsy.fr d'après Baskaren et coll)

Le rôle majeur des protéines Atg18/WIPI1-4, étudiées dans un chapitre spécial, est de transmettre les signaux PI(3)P aux protéines ATG en aval, en s'ancrant sur la membrane (

(Figure : vetopsy.fr d'après Dooley et coll)
2. Comme les sites de liaison avec l'Atg2/ATG2 par WIPI4 et de liaison Atg16L1 par WIPI2 sont situés à la surface opposée du motif FRRG, cette architecture permet aux Atg18/WIPI d'interagir simultanément avec les protéines ATG en aval.
3. Par ailleurs, la formation du complexe avec l'Atg2 et l'oligomérisation peuvent stabiliser davantage l'association membranaire des protéines Atg18/WIPI.
Remarque : ATG18 peut avoir un rôle distinct dans la fission de la membrane vacuolaire (
DFPC1
DFCP1 (double FYVE-containing protein 1, appelée aussi ZFYVE1) est une protéine de liaison à PI(3)P qui se localise aux omégasomes, i.e. le domaine FYVE, domaine zinc finger, et interagit avec les membranes cellulaires (Biogenesis of omegasomes and autophagosomes in mammalian autophagy 2024).
1. DFCP1 est une ATPase qui est activée par la liaison membranaire et se dimérise de manière dépendante de l’ATP (ATPase activity of DFCP1 controls selective autophagy 2023).
- DFCP1 serait indispensable et se localiserait au niveau des omégasomes, tout au long du processus de formation d'autophagosomes (Autophagosome formation from membrane compartments enriched in phosphatidylinositol 3-phosphate and dynamically connected to the endoplasmic reticulum 2008).
- La fonction de DFCP1 est inconnue, tout comme les mécanismes de formation et de constriction des omégasomes.

(Figure : vetopsy.fr d'après Nähse et coll)
2. Alors que la déplétion de DFCP1 a un effet mineur sur le flux autophagique en vrac, DFCP1 est nécessaire pour maintenir le flux autophagique de p62 dans l'autophagie sélective, dans des conditions d’alimentation et de dénutrition, et cela dépend de sa capacité à lier et à hydrolyser l’ATP.
a. Lors de la production de PI(3)P au niveau de l'omégasome, DFCP1 est formé de novo et on assiste au recrutement de p62 et LC3B quelques secondes plus tard en formant une sorte d'anneau.
Le phagophore forme une poche en se développant, et l'activité ATPase de DFCP1 est requise pendant la constriction de l'omégasome.
b. Dans les cellules exprimant le DFCP1 sauvage, l'omégasome se contracte rapidement.
En revanche, dans les cellules exprimant les mutants de l'ATPase de DFCP1, la constriction est inhibée, retardant la fermeture des omégasomes.
3. En outre, le DFCP1 régule la biogenèse des gouttelettes lipidiques (PI(3)P and DFCP1 regulate the biogenesis of lipid droplets 2022).
Les gouttelettes lipidiques (LD) sont impliquées dans la formation d'autophagosomes, ce qui implique que, dans certaines conditions, les gouttelettes lipidiques peuvent favoriser l'expansion du phagophore dans un processus coordonné par DFCP1 (Mammalian Atg2 proteins are essential for autophagosome formation and important for regulation of size and distribution of lipid droplets 2012).

Le turn-over de PI(3)P semble nécessaire pour la dissociation de la machine ATG de la surface des autophagosomes avant leur fusion avec le lysosome/vacuole (
Élongation du phagophore
Biologie cellulaire et moléculaireConstituants de la celluleSystème endomembranaireRéticulum endoplasmiqueAppareil de GolgiEndosomesLysosomesPeroxysomesAutophagieMacroautophagieMicroautophagieAutophagie chaperonnes (CMA)Autophagie non canoniqueProtéines ATGSystème UPS (Ubiquitine-Protéasome)Transport membranaireTrafic vésiculaireFusion/fission membranaireEndocytoseVoie sécrétoireMoteurs moléculairesVoies de signalisation


