Enzymes
Groupe des
transférases (EC 2)
Lipides kinases
Phosphatidylinositol Kinases (PI-kinases)
Phosphatidylinositol phosphates kinases : PIPKIII (PIKfyve/Fab1)
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Enzymes
- Vue d'ensemble
- Classification des enzymes
- Oxydoréductases (EC 1)
- Transférases (EC 2)
- Hydrolases (EC 3)
- Lyases ou synthases (EC4)
- Isomérases (EC5)
- Ligases ou synthétases (EC 6)
- Coenzymes
- Vitamines
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les phosphatidylinositol phosphates kinases (PIPK) sont une famille de kinases, phosphatidylinositol kinases (PIK ou PI-Kinases) capables de phosphoryler le groupe hydroxyle en position 4 ou en position 5 du cycle inositol des phosphoinositides.
Les PIPK produisent :
- du PI(4,5)P2 ou PIP2 :
- par la phosphorylation de PI(4)P par la classe I (PIPKI),
- par la phosphorylation de PI(5)P par la classe II (PIPKII) ;
- du P(3,5)P2 par la phosphorylation de PI(3)P pour la classe III (PIPKIII), PIKfyve des mammifères.
Vue d'ensemble des PIPKIII
p2.jpg)
(Figure : vetopsy.fr)
1. Les PIPK de classe III (EC 2.7.1.150) comprend un seul membre chez les vertébrés, PIKfyve, appelé aussi PIP5K3, homologue de Fab1p chez la levure, qui joue un rôle essentiel dans le système endosomal et lysosomal.
2. Fab1/PIKfyve est une phosphatidylinositol phosphates kinases (PIPK) et une phosphatidylinositol 5-kinases (PI5K) car il catalyse principalement la production de (Plentiful PtdIns5P from scanty PtdIns(3,5)P2 or from ample PtdIns? PIKfyve-dependent models: Evidence and speculation 2015) :
- PI(3,5)P2, en phosphorylant PI(3)P, en utilisant le pool créé par PI3KCIII/Vps34,
- mais aussi PI(5)P, en phosphorylant le phosphatidylinositol (PtdIns).
$\ce{ATP + PI(3)P}$ $\leftrightharpoons$ $\ce{ADP + PI(3,5)P2}$
$\ce{ATP + PtdIns}$ $\leftrightharpoons$ $\ce{ADP + PI(5)P}$

Pour plus de détails, reportez-vous à : PIKfyve and its Lipid Products in Health and in Sickness (2012) et PI5P and PI(3,5)P2: Minor, but Essential Phosphoinositides (2017).
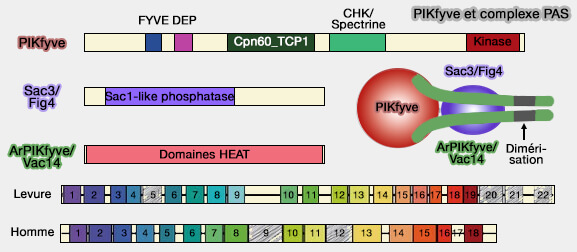
Structure de PIKfyve
Fab1/PIKfyve, contrairement aux autres classes de PIPK, est une longue protéine, formée d'environ 2050-2100 résidus qui comprend plusieurs domaines.
1. Le domaine N-terminal est formé par :
- un domaine FYVE (résidus 164-230), domaine de liaison à PI(3)P, essentiel pour localiser la protéine sur le feuillet cytosolique des endosomes,
- un domaine DEP (résidus 376-449), impliqué dans la voie dans les voies de signalisation des protéines G.
(Figure : vetopsy.fr)
2. Le domaine central (résidus 560-1438) est occupé par :s
- un domaine TCP1 (Teosinte branched 1 du maïs, Cycloidea du muflier à grandes fleurs, et PCF du riz), appelé aussi CCT (Chaperone Containing TCP1) qui peut se lier à Rab9 et à JLP, adaptateur de de protéines motrices, impliquées tous deux dans le transport des endosomes vers le réseau trans-Golgi (TGN),
- un domaine CHK, riche en cystéine, histidine et lysine, appelé aussi CCR (Conserved Cysteine Rich) dont la région lysine est homologue des répétitions de spectrine trouvées dans de nombreuses protéines liées au cytosquelette.
Ce domaine central interagit avec de nombreux partenaires de Fab1/PIKfyve.
3. Le domaine catalytique (résidus 1801–2038) montre de nombreuses similitudes avec les domaines kinase de PIPKI et PIPKII, mais pas avec ceux des PI3K et des PI4K.
- La lysine en position 1831 est responsable de la liaison avec l'ATP.
- La lysine 1999 a une préférence pour PI(3)P alors que la lysine 2000 a une préférence pour le phosphatidylinositol (Functional Dissection of Lipid and Protein Kinase Signals of PIKfyve Reveals the Role of PtdIns 3,5-P2 Production for Endomembrane Integrity 2002).
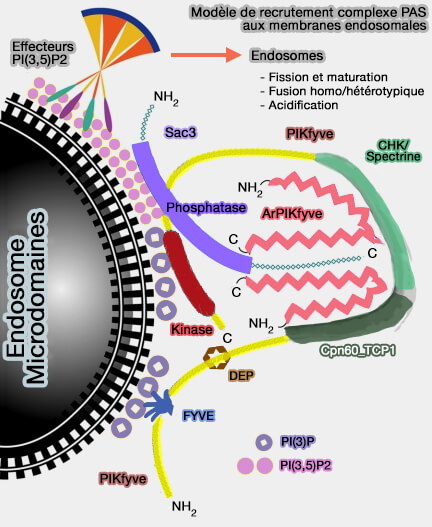
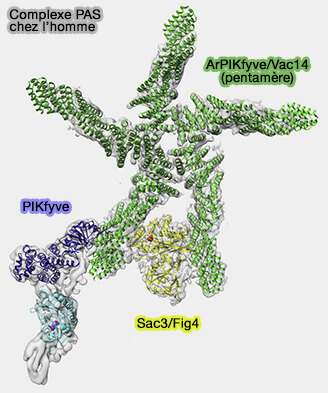
Complexe PAS
Fab1/PIKfyve est retrouvé sous forme de complexe (PAS), complexe qui régule la synthèse de PI(3,5)P2 et de PI(5)P, dans les endosomes précoces et tardifs et les lysosomes, et qui comprend deux autres protéines :
- Vac14/ArPIKfyve (Associated Regulator of PIKfyve), A de PAS,
- Sac3/Fig4, S de PAS.

Pour plus de détails, reportez-vous à : Insights into Lysosomal PI(3,5)P2 Homeostasis from a Structural-Biochemical Analysis of the PIKfyve Lipid Kinase Complex (2020) et Phosphatidylinositol 3,5-bisphosphate: Regulation of cellular events in space and time (2016).
Vac14/ArPIKfyve
1. Vac14/ArPIKfyve est une protéine de 782 résidus qui comprend des domaines de liaison protéine/protéine (PIKfyve-ArPIKfyve-Sac3 Core Complex : contact sites and their consequence for Sac3 phosphatase activity et endocytic membrane homeostasis 2009).
(Figure : vetopsy.fr d'après Ikomonov et coll)
- Vac14/ArPIKfyve devrait être entièrement composé de répétitions HEAT (18 à 22 chez la levure 15 à 17 répétitions HEAT chez l'homme, chacune contenant deux hélices antiparallèles reliées par une boucle courte.
- Certains auteurs trouvent aussi des domaines coiled-coil (superhélice), en particulier C-terminaux.
2. Ces motifs permettent dans l'ordre (Vac14 Protein Multimerization Is a Prerequisite Step for Fab1 Protein Complex Assembly and Function 2013) :
- l'homodimérisation de Vac14/ArPIKfyve,
- la liaison avec Sac3/Fig4,
- la liaison avec Fab1/PIKfyve (Biallelic Mutations of VAC14 in Pediatric-Onset Neurological Disease 2016).
3. Vac14/ArPIKfyve est nécessaire à la phosphorylation de PI(3)P et du phosphatidylinositol (PtdIns) par Fab1/PIKfyve (ArPIKfyve homomeric and heteromeric interactions scaffold PIKfyve and Sac3 in a complex to promote PIKfyve activity and functionality 2008).
4. En outre, Vac14/ArPIKfyve pourrait se lier à Rab9 et Rab7, régulateurs de la dynamique de la membrane endo-lysosomale et des voies de trafic (The Vac14-interaction Network Is Linked to Regulators of the Endolysosomal and Autophagic Pathway 2014).
5. Une étude récente montre que chez l'homme, Vac14/ArPIKfyve se présenterait sous forme de pentamère en forme d'étoile qui peut se lier qu'à une seule copie de PIKfyve et de Fig4 (Insights into Lysosomal PI(3,5)P2 Homeostasis from a Structural-Biochemical Analysis of the PIKfyve Lipid Kinase Complex 2020).
Sac3/Fig4
1. Sac3/Fig4, une phosphatase qui ne se trouve que dans ce complexe et qui appartient aux phosphatases SAC, i.e. phosphatases non spécifiques (EC 3.1.3.25) contenant un domaine Sac, qui hydrolyse le phosphate en position 3, 4 ou 5.
$\ce{PIP + H2O}$ $\leftrightharpoons$ $\ce{PI + phosphate}$
Sac3/Fig4, a un rôle inhabituel par l'activation de la kinase Fab1/PIKfyve.
- Il est nécessaire à l'activité kinase du complexe.
- Il agit agir en tant que phosphatase active dans le complexe.
(Figure : vetopsy.fr d'après Lees et coll)
2. Le rôle de Sac3/Fig4 est énigmatique (Insights into Lysosomal PI(3,5)P2 Homeostasis from a Structural-Biochemical Analysis of the PIKfyve Lipid Kinase Complex 2020).
- Il semblerait que la conformation du complexe empêcherait le domaine kinase de Fab1/PIKfyve d'accéder au PI(3)P membranaire, et donc de produire PI(3,5)P2.
- Sac3/Fig4 pourrait amorcer une activité maximale suite à un changement de conformation, tel qu'une dissociation complexe, en réponse à un stimulus encore inconnu.
Ce modèle expliquerait pourquoi l'ablation de l'activité Sac3/Fig4 déprime la production de PI(3,5)P2 dans les cellules, mais ?
3. PAS est parcouru par deux entrées antagonistes :
- une pour la synthèse de PI(3,5)P2,
- une autre pour son turn-over pour obtenir du PI(3)P ou même, du phosphatidylinositol (PtdIns) par Sac3/Fig4.
Un tel cycle de phosphorylation-déphosphorylation élève le flux de PI(3,5)P2, entraînant sa diffusion réduite et sa disponibilité locale accrue. Par contre, la régulation de ces processus n'est pas encore identifiée.
4. Les déficiences en Sac3/Fig4 provoquent de nombreuses maladies comme la maladie de Charcot de type-4J (Severe Consequences of SAC3/FIG4 Phosphatase Deficiency to Phosphoinositides in Patients with Charcot-Marie-Tooth Disease Type-4J 2019).
Autres composants ?
Chez la levure, l'interconversion dynamique de PI(3)P et PI(3,5)P2 est régulée par au moins cinq protéines (VAC14 nucleates a protein complex essential for the acute interconversion of PI3P and PI(3,5)P2 in yeast and mouse 2008).
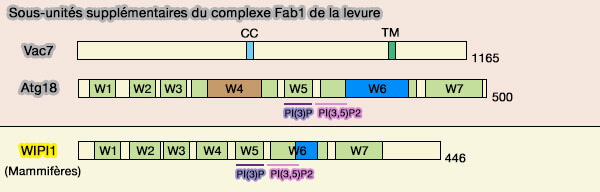
(Figure : vetopsy.fr)
1. Fab1/PIKfyve est la phophatidylinositol 5-kinase.
2. Sac3/Fig4 est une phosphatase qui agit sur la kinase Fab1/PIKfyve
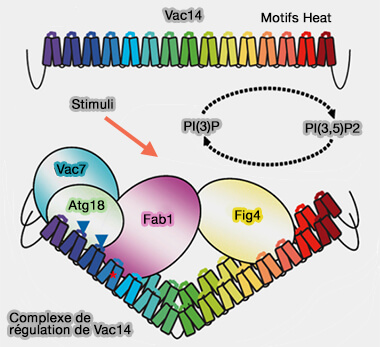
(Figure : vetopsy.fr d'après Jin et coll)
3. Vac14/ArPIKfyve, qui stimule l'activité de la kinase, sert de plaque tournante au complexe et, par le biais de répétitions HEAT distinctes, lie directement Fab1/PIKfyve, Sac3/Fig4, Vac7 et Atg18/WIPI.
4. Vac7, une autre sous-unité du complexe de la levure, stimule l'activation de Fab1/PIKfyve .
- Vac7 est impliqué dans la scission de la membrane vacuolaire et nécessaire pour l'acidification et la morphologie normales des vacuoles (Vac7p, a Novel Vacuolar Protein, Is Required for Normal Vacuole Inheritance and Morphology 1997).
- Les homologues de Vac7 n'ont été observés que chez les champignons.
Sac3/Fig4, la lipide phosphatase, possède le même double rôle (cf. plus haut).
5. Atg18 régule négativement l'activité de Fab1/PIKfyve.
Chez les mammifères, Atg18 a quatre homologues PROPPIN, WIPI (WD-repeat protein Interacting with PhosphoInositides)-1, 2, 3 et 4.

Atg8 et les WIPI sont étudiés dans un chapitre spécifique et ont un rôle majeur dans la formation des autophagosomes (
Rôles de Fab1/PIKfyve
1. Fab1/PIKfyve joue un rôle essentiel dans le processus de maturation des endosomes en régulant (PIKfyve and its Lipid Products in Health and in Sickness 2012) :
- l'homéostasie membranaire,
- le trafic endosomal, i.e. des endosomes précoces vers les endosomes tardifs, des cargos et des protéines membranaires destinés à la dégradation lysosomale, ou vers le réseauréseau trans-Golgi (TGN),
- l'autophagie.
2. Fab1/PIKfyve a un rôle fondamental dans la fission lysosomale (
L'altération de Fab1/PIKfyve provoque une hypertrophie des lysosomes et une vacuolisation cytoplasmique en raison du défauts de synthèse de PI(3,5)P2 et de fission des lysosomes

Les endosomes et le système endomembranaire sont traités dans des chapitres spécifiques.
2. Fab1/PIKfyve joue un rôle clé dans de multiples processus biologiques, tels que la translocation de GLUT4 et le bourgeonnement rétroviral.

Fab1/PIKfyve devient une cible pour le cancer, les infections virales et les maladies neurodégénératives (Small molecule PIKfyve inhibitors as cancer therapeutics: translational promises and limitation 2019).
Retour aux phosphatidylinositol kinases (PIK ou PI-Kinases)
BiochimieChimie organiqueBioénergétiqueProtidesGlucidesLipidesEnzymesKinasesProtéines kinasesLipides kinasesHydrolasesPeptidases/protéasesPhosphatasesEstérasesLipasesPhospholipasesATPasesGTPasesProtéines GPetites GTPasesFamille dynamineCoenzymesVitaminesHormonesComposés inorganiquesTransport membranaireMoteurs moléculaires


