Système Ubiquitine-Protéasome (UPS)
Protéasome 26S
Vue d'ensemble et structure générale
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les protéasomes sont des complexes enzymatiques multiprotéiques dont le rôle est de découper les protéines (protéolyse) en peptides.
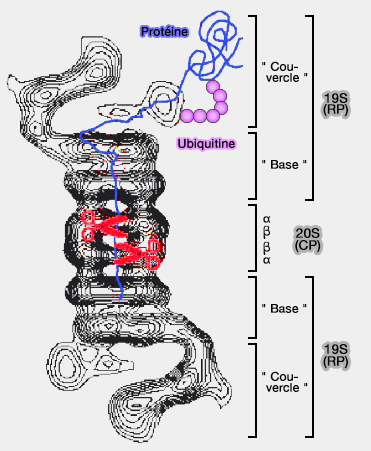
(Figure : vetopsy.fr d'après Tanaka)
Les protéasomes sont essentiels à de nombreux processus cellulaires tels que le cycle cellulaire, la réparation de l'ADN, l'apoptose, la transduction des signaux, la réponse immunitaire, le développement embryonnaire et le contrôle de la qualité des protéines (The proteasome: Overview of structure and functions 2009).
Jusqu'en 1977, on pensait que la dégradation des protéines ne s'effectuait que dans les lysosomes, organites à membrane entourant un contenu acide riche en protéases. La découverte de l'ubiquitine et l'identification du protéasome valut à Irvin Rose, Avram Hershko et Aaron Ciechanover le prix Nobel de chimie en 2004 (cf. historique).
Vue d'ensemble
Ces protéasomes sont déjà présents chez les Archées, certaines bactéries (Actinomycètes) et dans toutes les cellules eucaryotes (Catalytic Mechanism and Assembly of the Proteasome 2009).
- Les protéasomes des espèces primitives correspondent uniquement à la particule 20S du protéasome 26S des Eucaryotes (Prokaryotic Proteasomes: Nanocompartments of Degradation 2013).
- Les bactéries, à l'exception des Actinomycètes, ne possèdent pas de protéasome 20S, mais un complexe analogue ATP-dépendant en forme de tonneau - par exemple, de la famille Lon (Functional mechanics of the ATP-dependent Lon protease- lessons from endogenous protein and synthetic peptide substrates 2008) ou de la famille Clp, responsable de 80% de la dégradation des protéines chez Escherichia coli -.
- Ces complexes se retrouvent aussi chez les eucaryotes (Human Mitochondrial ClpP Is a Stable Heptamer That Assembles into a Tetradecamer in the Presence of ClpX 2005).
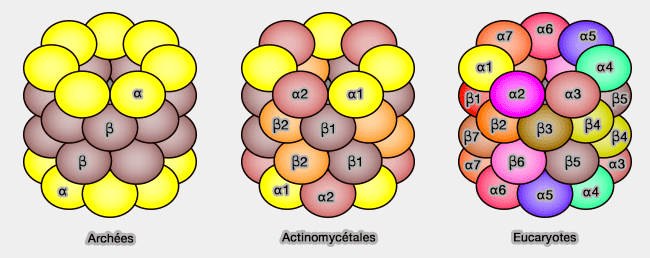
(Figure : vetopsy.fr)
Le système ubiquitine-protéasome (UPS) est la principale voie de dégradation ATP-dépendante des protéines régulatrices, ou mal pliées et anormales dans le cytosol et le noyau des cellules eucaryotes. Le protéasome 26S en est un élément central (Regulated protein turnover: snapshots of the proteasome in action 2014 et tout savoir sur l'UPS de Saccharomyces cerevisiae : The Ubiquitin–Proteasome System of Saccharomyces cerevisiae 2012).
-
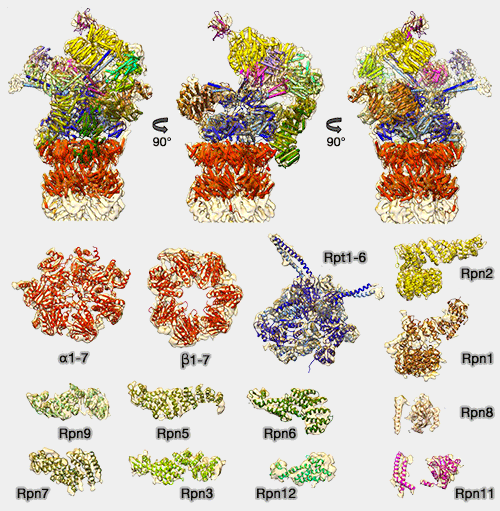
Protéasome 26S et ses sous-unités
(Figure : vetopsy.fr d'après Beck) - D'autres le sont indépendamment de ce marquage, comme Rpn4 - régulateur transcriptionnel du protéasome -, la thymidylate syntase - production de précurseurs de l'ADN - et l'ornithine-décarboxylase - biosynthèse des polyamines - (Ubiquitin-independent proteasomal degradation 2014).
Une fois l'acceptation du substrat effectuée, le processus est lancé et le protéasome :
- déplace la chaîne polypeptidique du substrat (cf. engagement du substrat), hydrolysant l'ATP en ADP (cycle ATPasique) dans les Rpt de la base de la particule 19S pour déplier et transloquer les protéines vers la chambre catalytique (cf. translocation du substrat),
- clive l'ubiquitine qui peut être alors recyclée (cf. désubiquitination du substrat),
- coupe la protéine séquentiellement en peptides courts de 7 à 8 acides aminés (cf. protéolyse du substrat).
Ce processus complexe lui permet :
- de remodeler des complexes protéiques en dégradant que la sous-unité sur laquelle il initie la première dégradation, par exemple, en clivant la partie active des protéines-cibles ou un domaine activateur ou inhibiteur dans les voies de signalisation par exemple ;
- de dégrader les protéines.
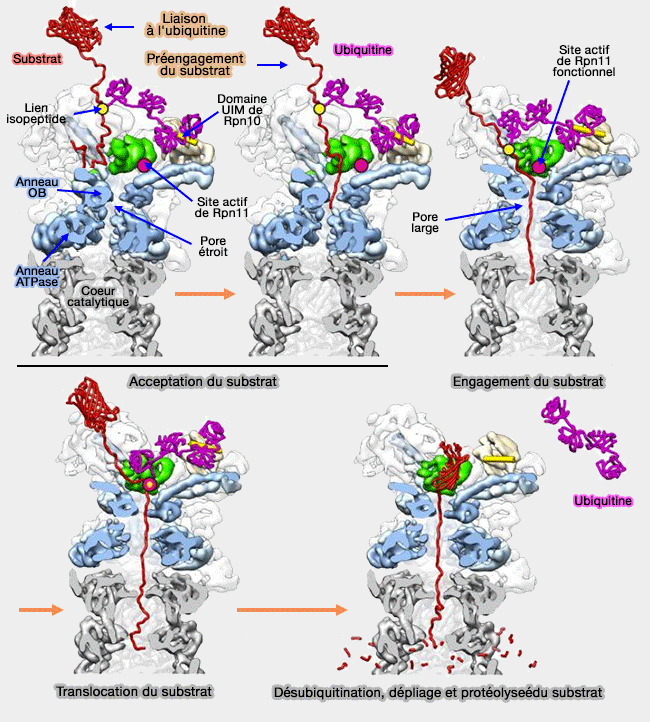
(Figure : vetopsy.fr d'après Lander)
Les pages qui suivent seront surtout axées sur le protéasome 26S.
Structure générale du protéasome 26S
Les composants du protéasome sont nommés d'après leur coefficient de sédimentation de Svedberg (S).
Vous pouvez aussi voir un film sur la structure générale tiré de l'article : Near-atomic resolution structural model of the yeast 26S proteasome 2012.
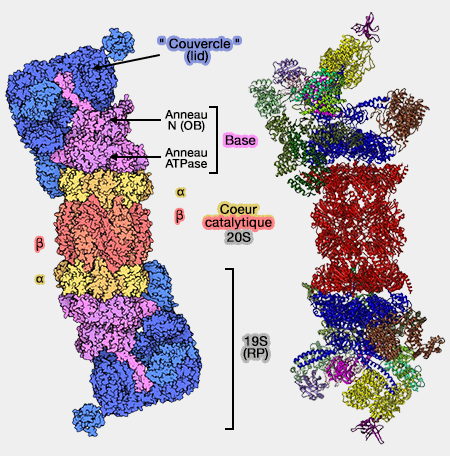
(Figure : vetopsy.fr)
Le protéasome 26S, de masse moléculaire 2000 kDa, est le plus utilisé par les Mammifères (Molecular architecture of the 26S proteasome holocomplex determined by an integrative approach 2012).
Il est formé de 33 protéines différentes et contient :
- un cœur catalytique 20S (CP : Core Particle) en forme de tonneau, le 20S, creux, à 4 anneaux heptamériques, dans lequel les protéines pénètrent par un pore étroit et sont plus ou moins dégradées (cf. protéolyse du substrat) ;
- deux complexes régulateurs (RP : Regulatory Particle) en règle générale, à activité ATPasique, placés de chaque côté de celui-ci, contenant des sites actifs et des sites de liaison à l'ubiquitine pour la reconnaissance des protéines polyubiquitinées (cf. acceptation du substrat) et leur transfert vers la chambre catalytique (cf. engagement et translocation du substrat).
Le protéasome est une structure dynamique qui modifie sa conformation durant le processus et qui interagit avec un grand nombre de protéines.
Coeur catalytique (CP : Core Particle) : particule 20S
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


