Enzymes
Groupe des
transférases (EC 2)
Lipides kinases
Phosphatidylinositol Kinases (PI-kinases)
Phosphatidylinositol 3-kinases (PI3K) : PI3KC3-CI
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Enzymes
- Vue d'ensemble
- Classification des enzymes
- Oxydoréductases (EC 1)
- Transférases (EC 2)
- Hydrolases (EC 3)
- Lyases ou synthases (EC4)
- Isomérases (EC5)
- Ligases ou synthétases (EC 6)
- Coenzymes
- Vitamines
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

PIK3C3 forme à lui seul la classe III de la une famille des PI3K, kinases, phosphatidylinositol kinases (PIK ou PI-Kinases) capables de phosphoryler le groupe hydroxyle en position 3 du cycle inositol des phosphoinositides.
PIKC3, Vps34 chez la levure, forment deux complexes :
- PI3KC3-CI impliqué dans la formation des autophagosomes,
- PIKC3-CII localisé dans les membranes endosomales.
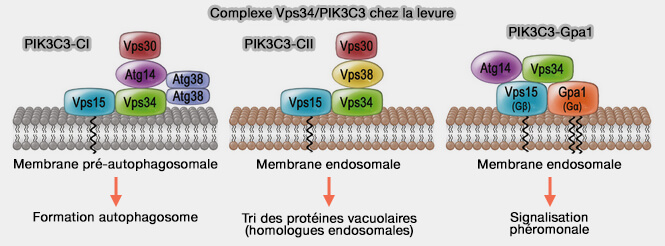
(Figure : vetopsy.fr d'après Reidick et coll)
PI3KC3-CI
Dans le complexe PI3KC3-CI, Vps34 est responsable de la production de PI(3)P sur les membranes impliquées dans la formation des autophagosomes, appelées membranes pré-autophagosomales (La formation de l'autophagosome 2017).
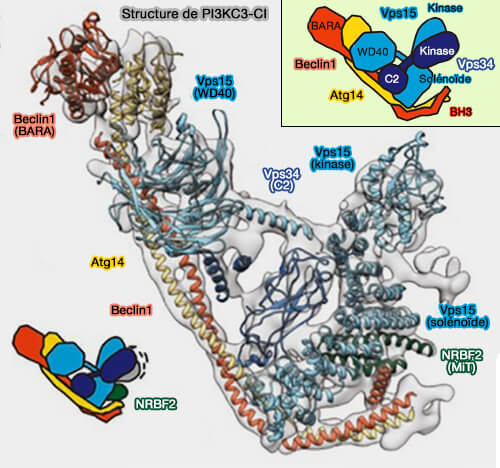
Structure du complexe
PI3KC3-CI, outre Vps34, Vps15/p150 et Vps30/Beclin1(BECN) comprend deux autres protéines, appelées ATG (AuTophaGy-related protein) :
- Atg14/ATG14L
- Atg38/NRBF2.

Vous pouvez lire : Emerging roles of ATG proteins and membrane lipids in autophagosome formation (2020) et Phosphoinositide 3-kinases as accelerators and brakes of autophagy (2013) et Structural pathway for allosteric activation of the autophagic PI 3-kinase complex I (2019).

(Figure : vetopsy.fr d'après Nishimura et Tooze)
Atg14
Atg14/ATG14 ou BARKOR est indispensable pour la localisation et l'assemblage appropriés de la machinerie autophagique spécifique sur le site, car elle recrute Atg5 et Atg8 dans les membranes de la structure pré-autophagosomale (PAS ou Pre-Autophagosomal Structure), qui contiennent également ULK/Atg1, Atg2 et Atg16 (Membrane-binding domains in autophagy 2019).
Structure d'ATG14
Atg14 possèdent plusieurs domaines.
(Figure : vetopsy.fr d'après Young et coll)
1. Les répétitions N-terminales de cystéine d’ATG14 sont essentielles pour la localisation du RE (Autophagy requires endoplasmic reticulum targeting of the PI3-kinase complex via Atg14L 2009).
La phosphorylation de S29 dans l’extrémité N-terminale d’ATG14 par ULK1/2 est d’une importance vitale pour l’activation de l’activité de la kinase lipidique du complexe PI3KC3-C1 à la fois lors d’une privation d’acides aminés, d’une privation de glutamine et pendant l’hypoxie (ULK1 phosphorylates Ser30 of BECN1 in association with ATG14 to stimulate autophagy induction 2018).
2. Le domaine BATS (Barkor/Atg14(L) Autophagosome-Targeting Sequence) à son extrémité C-terminale est responsable :
- du ciblage des autophagosomes,
- de la détection de la courbure de la membrane,
- de la liaison directe aux membranes par le biais d’une hélice alpha amphipathique. qui se lie aux membranes fortements incurvées (Autophagosome targeting and membrane curvature sensing by Barkor/Atg14(L) 2011 et Membrane-binding domains in autophagy 2019),
Le domaine BATS se lie à la fois à P(I)3P et à PI(4,5)P2.
Son interaction avec PI(4,5)P2 régule l’interaction d’ATG14 avec PIK3C3 et BECN1, influençant l’assemblage complexe et l’initiation de l’autophagie (PtdIns(4,5)P2 signaling regulates ATG14 and autophagy 2016)
3. Deux domaines LIR seraient présents dont un domaine proche du domaine BATS, i.e. BATS et LIR pourraient fonctionner comme un détecteur de coincidence pour le ciblage spécifique des membranes autophagiques.
VPS34 et BECLIN1 contiennent également des motifs LIR fonctionnels et interagissent préférentiellement avec GABARAP et GABARAPL1 (Members of the autophagy class III phosphatidylinositol 3-kinase complex I interact with GABARAP and GABARAPL1 via LIR motifs 2019).

(Figure : vetopsy.fr d'après Nishimura et Tooze)
Rôles d'ATG14
ATG14 aurait plusieurs rôles.
1. Son rôle dans la nucléation du phagophore a été étudié plus haut et celui, plus général de PI3KC3-C1, est étudié dans le chapitre idoine (
2. ATG14 a été impliqué dans la fusion autophagosome-endolysosome (ATG14 promotes membrane tethering and fusion of autophagosomes to endolysosomes 2015).
a. La majeure partie de l’ATG14 ou du complexe PI3KC3-C1 se dissocie de la membrane du phagophore avec d’autres ATG au cours de la formation des autophagosomes (Temporal analysis of recruitment of mammalian ATG proteins to the autophagosome formation site 2013).
Il est possible qu’une petite fraction reste sur la membrane et qu'après dissociation de la membrane du phagophore, ATG14, probablement sans autres composants de PI3KC3, est re-recruté dans les autophagosomes matures pour faciliter la fusion autophagosome-lysosome, sans que l'on sache comment.
b. Sur les autophagosomes, ATG14 :
- se lie au domaine central SNARE de STX17 par le biais de son domaine coiled-coil,
- stabilise le complexe binaire t-SNARE STX17-SNAP29 sur les autophagosomes et le prépare à l’interaction avec VAMP8, localisé dans l’endolysosome pour favoriser la fusion membranaire entre les autophagosomes et les endolysosomes.
c. Les activités de liaison à STX17, d’attache membranaire et d’amélioration de la fusion d’ATG14 nécessitent son homo-oligomérisation par ses répétitions de cystéine.
Dans les cellules ATG14 défectueuses d’homo-oligomérisation, les autophagosomes se forment toujours efficacement mais leur fusion avec les endolysosomes est bloquée.
Atg38/NRBF2
1. Atg38/NRBF2, entre en interaction avec Vps34 et Atg14 par son domaine MIT N-terminal (NRBF2 regulates macroautophagy as a component of Vps34 Complex I 2015)
- Le domaine MIT est localisé à la base du bras droit, à l'arrière du complexe dans la vue standard.
- MIT interagit fortement avec la partie centrale du domaine hélicoïdal de VPS15, les portions N-terminales de Beclin1 et Atg14.
Atg38/NRBF2 pourrait :
- stabiliser les interactions (Ubiquitination and phosphorylation of Beclin 1 and its binding partners: Tuning class III phosphatidylinositol 3-kinase activity and tumor suppression 2012),
- améliorer l'activité de VPS34, même si son rôle est controversé (Dynamics and architecture of the NRBF2-containing phosphatidylinositol 3-kinase complex I of autophagy 2016).
- être le lien qui incorpore le complexe CI aux réseaux de signalisation ou aux membranes (Characterization of Atg38 and NRBF2, a fifth subunit of the autophagic Vps34/PIK3C3 complex 2016).
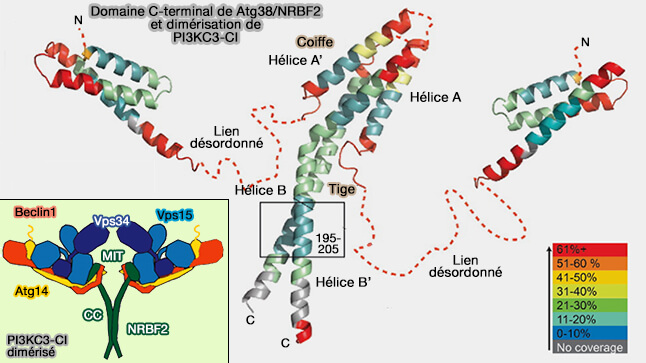
(Figure : vetopsy.fr d'après Ohashi et coll et Young et coll)
2. Atg38/NRBF2 dimérise PI3KC3-CI via son domaine C-terminal pour former des complexes dodécamères (Characterization of Atg38 and NRBF2, a fifth subunit of the autophagic Vps34/PIK3C3 complex 2016 et Dynamics and architecture of the NRBF2-containing phosphatidylinositol 3-kinase complex I of autophagy 2016).

La dynamique intra-domaine joue un rôle clé pour réguler l'activité de Vps34 car les domaines hélicoïdal et kinase, par leur flexibilité, peuvent faire adopter à PI3KC3-CI plusieurs conformations (Vps34 Kinase Domain Dynamics Regulate the Autophagic PI 3-Kinase Complex 2017).
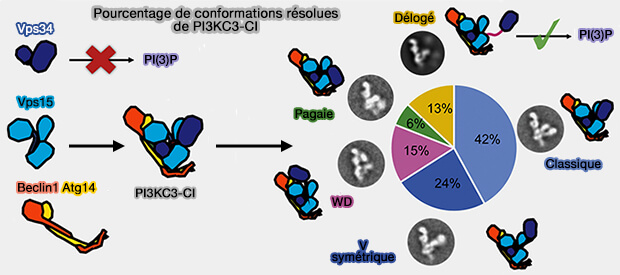
(Figure : vetopsy.fr d'après Sjepanovic et coll)
Ciblage de la membrane
Le ciblage de PIK3C3-CI vers la membrane du RE est facilité par plusieurs sous-unités qui forment deux bras :
-

PI3KC3-CI et contacts membranaires
(Figure : vetopsy.fr d'après Melia et coll) - un domaine riche en cystéine N-terminal et un domaine BATS dans ATG14,
- le domaine C-terminal, domaine hélicoïdal et lipide-kinase de Vps34,
- le domaine N-terminal de myristoylation de VPS15.
1. La région en boucle C-terminale peut interagir avec les membranes et permettre l'activation de Vps34 (Structure and flexibility of the endosomal Vps34 complex reveals the basis of its function on membranes 2016).
L'extrémité C-termonale de VPS34 est régulée par l'acétylation de K771, ce qui entrave l'affinité du VPS34 pour son substrat PI et son association avec BECN1 (VPS34 Acetylation Controls Its Lipid Kinase Activity and the Initiation of Canonical and Non-canonical Autophagy 2017).
2. Les résidus aromatiques dans une boucle de surface du domaine BARA du Vps30/BECLIN1 servent de doigt hydrophobe pour servir d'association directe avec les membranes.
3. La géométrie du site catalytique VPS34 est fortement influencée par la présence de VPS15, indiquant que le VPS15 joue un rôle central dans l'assemblage complexe d'échafaudage (Vps34 kinase domain dynamics regulate the autophagic PI 3-kinase complex 2017).
4. Le domaine BATS se lie aux petits liposomes contenant PI3P ou PI(4,5)P2 et cible la courbure de la membrane par l'intermédiaire d'une boucle d'hélice amphatique.

L'extrémité C-terminale de VPS34 détermine l'orientation du complexe PI3KC3-C1, tandis que le domaine BATS ATG14L1 est essentiel pour détecter la courbure de la membrane et médier la spécificité des lipides aux membranes cibles (Cryo-EM structure and biochemical analysis reveal the basis of the functional difference between human PI3KC3-C1 and -C2 2017).
Rôle de Vps34 dans l'autophagie
En outre, il semblerait que Vps34 pourrait réguler négativement l'autophagie par plusieurs processus.
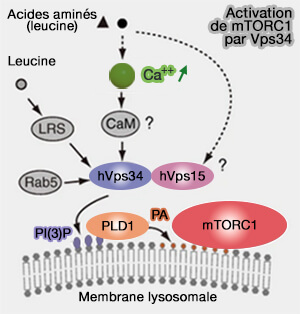
(Figure : vetopsy.fr d'après Takahra et coll)
1. Vps34 détecterait les acides aminés par l'augmentation du taux de Ca++ intracellulaire qui conduit à la liaison directe de la Ca++/calmoduline (CaM) à Vps34, ce qui déclenche la production de PI(3)P à partir du phosphatidylinositol (Amino Acids Activate mTOR Complex 1 via Ca2+/CaM Signaling to hVps34 2008).
Ce processus fait débat car certains auteurs pensent que c'est Vps15 qui, rappelons-le possède un domaine protéine kinase, dont on ne sait pas s'il est actif ou pas (pseudokinase), même si Vps34 peut se lier à la CAM (hVps15, but not Ca2+/CaM, is required for the activity and regulation of hVps34 in mammalian cells 2009)
2. L'augmentation de PI(3)P par Vps34 recrute la phospholipase D1 (PLD1), via son domaine PX, dans les lysosomes, ce qui provoque la production de d'acide phosphatidique (PA) et l'activation de mTORC1 (Class III PI-3-kinase activates phospholipase D in an amino acid–sensing mTORC1 pathway 2011 et Amino acid-dependent control of mTORC1 signaling: a variety of regulatory modes 2020).
3. Rab5A, régulateur de la production de PI(3)P par Vps34 est probablement impliquée dans l'activation de mTORC1 (Rab5 Proteins Regulate Activation and Localization of Target of Rapamycin Complex 1 2012),
En plus, mTORC1 :
- inhibe spécifiquement l'autophagie en favorisant PI3KC3-CI (Regulation of PIK3C3/VPS34 complexes by MTOR innutrient stress-induced autophagy 2013),
- active PI3KC3-CII, via la phosphorylation de Vps38/UVRAG pendant le processus de reformation autophagosome-lysosome après récupération lors de famine (mTOR activates the VPS34–UVRAG complex to regulate autolysosomal tubulation and cell survival 2015).

Par conséquent, les différents complexes Vps34 ont non seulement des effets opposés dans la cellule, mais peuvent potentiellement se réguler négativement.
PI3KC3-CII
BiochimieChimie organiqueBioénergétiqueProtidesGlucidesLipidesEnzymesKinasesProtéines kinasesLipides kinasesHydrolasesPeptidases/protéasesPhosphatasesEstérasesLipasesPhospholipasesATPasesGTPasesProtéines GPetites GTPasesFamille dynamineCoenzymesVitaminesHormonesComposés inorganiquesTransport membranaireMoteurs moléculaires


