Dégradation des protéines
Dégrons et signaux de reconnaissance
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les dégrons sont des signaux peptidiques ou structuraux qui régulent la stabilité des protéines en conditionnant leur reconnaissance, leur ubiquitination et leur dégradation par le système ubiquitine-protéasome (UPS).
Propriétés générales des dégrons
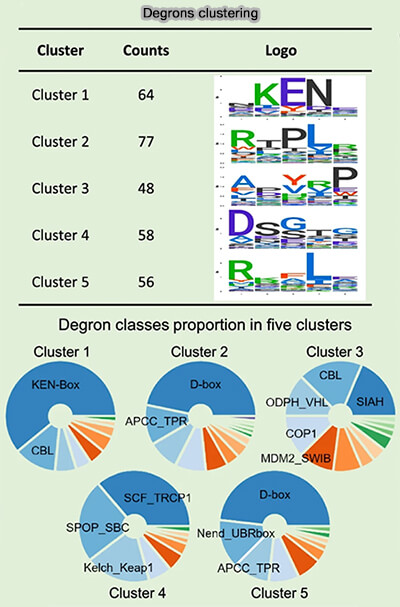
(Figure : vetopsy.fr d'après Hou et coll)
Les signaux de dégradation, généralement courts (3 à 15 acides aminés), sont reconnus par des composants du système ubiquitine-protéasome (UPS), notamment par des ligases E3, qui catalysent l’attachement de chaînes d’ubiquitine sur la protéine cible (Systematic prediction of degrons and E3 ubiquitin ligase binding via deep learning 2022).
1. On distingue plusieurs catégories selon leur nature et leur mode d’activation.
- Les dégrons constitutifs sont présents en permanence et assurent une dégradation continue.
- Les dégrons conditionnels sont exposés ou activés en réponse à un signal post-traductionnel comme la phosphorylation, le clivage protéolytique ou le démasquage conformationnel.
- Les dégrons contextuels dépendent de l’environnement cellulaire, i.e. pH, stress oxydatif, interaction protéique.
2. La reconnaissance d’un dégron implique :
- des éléments structuraux spécifiques, i.e. motif hydrophobe, polarité de charge, structure secondaire,
- des modifications activatrices, assurant la sélectivité des E3 ligases.
Classification des dégrons
La classification des dégrons est conditionnée par leur position dans la protéine et leur mode d’activation.

La classification est résumée dans le tableau suivant dont les liens renvoient aux pages spécifiques.
| Catégorie | Localisation / caractéristiques | Exemples et renvois |
|---|---|---|
| Dégrons terminaux |
|
|
| Dégrons internes | ||
| Dégrons internes (généralités) |
||
| Phospho dégrons |
|
|
| Dégrons conformationnels et structurels |
|
|
| Dégrons cycle cellulaire |
|
|
Dégrons constitutifs et conditionnels
On peut également classer les dégrons selon leur mode d’apparition.
1. Certains dégrons, dits conditionnels, ne sont pas présents dans la structure native des protéines, mais apparaissent uniquement après un événement biologique particulier.
- Les dégrons créés par clivage protéolytique exposent une nouvelle extrémité, comme lors de l’activation des caspases (
voie Arg/N-end rule).
- Les dégrons exposés par changement conformationnel font apparaître des régions hydrophobes reconnues par le système ERAD ou par les chaperonnes cytosoliques associées au protéasome.
- Les dégrons activés par des modifications post-traductionnelles incluent par exemple le Cdc4 phosphodégron (CPD) après phosphorylation de p27Kip1.
- Les dégrons masqués par interaction protéique deviennent accessibles après dissociation d’un complexe, comme pour β-caténine reconnue par SCFβ-TrCP.
2. D’autres dégrons sont contextuels, leur activation dépendant de signaux environnementaux ou de conditions physiologiques spécifiques.
- Les dégrons déclenchés par hydroxylation, phosphorylation ou acétylation assurent une régulation dépendante du contexte cellulaire, comme HIF1α reconnu par VHL après hydroxylation en condition normoxique.
- Les dégrons soumis à une ubiquitination régulée participent à des mécanismes de signalisation spécifiques, tels que Smad4 ubiquitiné par TRIM33 (ectodermine).
- Les dégrons exposés lors de stress oxydatif, mitochondrial ou protéotoxique permettent la reconnaissance des protéines endommagées par des E3 spécialisées, comme la parkine ou CHIP.
3. Ces mécanismes illustrent la capacité de la cellule à coupler les voies de signalisation, le repliement et le clivage à la dégradation sélective des protéines, garantissant une régulation fine et adaptative du protéome.
Dégrons hybrides et multisignaux
Certaines protéines possèdent plusieurs dégrons distincts, activés de manière coordonnée ou séquentielle.
1. Ces dégrons hybrides ou multi-signaux intègrent différents déterminants, i.e. structuraux, post-traductionnels ou conformationnels, pour ajuster finement la dégradation selon le contexte cellulaire et permettent la combinaison de voies de régulation.
- Un dégron interne ou terminal peut être modulé par une phosphorylation ou une acétylation, comme la cycline F, dont le dégron hydrophobe interne est régulé au cours du cycle cellulaire,
- Un motif conformationnel peut être exposé après activation enzymatique, comme la parkine, dont l’activation par la kinase Pink1 déclenche la mitophagie.
- L’association de plusieurs signaux reconnus par des E3 distinctes, comme le couplage entre phospho-dégrons et D-box, contrôle la dégradation séquentielle des régulateurs mitotiques.
2. Ces mécanismes illustrent la capacité du système ubiquitine-protéasome à intégrer plusieurs niveaux de signalisation pour contrôler avec précision la stabilité des protéines.
Reconnaissance des dégrons par les ligases E3
La reconnaissance des dégrons constitue un principe général de biologie cellulaire, déployé par des ligases E3 aux architectures diverses mais qui convertissent un signal moléculaire en décision de dégradation, reliant les propriétés intrinsèques du substrat à la machinerie d'ubiquitination.
Selon la nature du dégron, séquence terminale, motif interne, phosphorylation, conformation transitoire, interface structurale ou signal dépendant du cycle cellulaire, différentes ligases E3 utilisent des modules de liaison spécialisés pour assurer une sélection fine et hautement contextuelle.
1. Les E3 ubiquitine ligases sont classées en trois grandes familles (New insights into ubiquitin E3 ligase mechanism 2014 et HECT and RING finger families of E3 ubiquitin ligases at a glance 2012).
- la famille HECT,
- la famille RING, à domaine RING ou RING-related, les U-box ne contenant pas d'atomes de zinc y sont intégrées,
- la famille RBR (RING-Between-RING).

Le dégron agit comme une adresse moléculaire indiquant à la ligase E3 qu’un substrat doit être ubiquitiné.
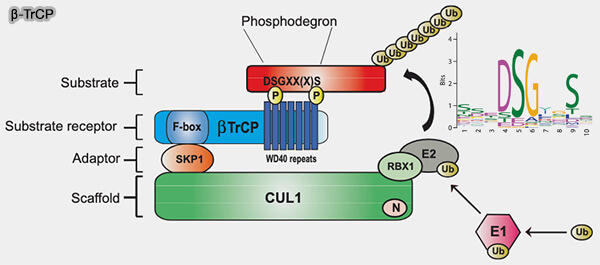
(Figure : vetopsy.fr d'après Low et coll)
2. Les principaux modes de reconnaissance incluent :
- la reconnaissance de séquences primaires par de nombreuses E3 activées en continu qui identifient des motifs définis, comme les extrémités N-terminales instables (N-end rule) ou les queues C-terminales via des domaines adaptateurs dédiés,
- la reconnaissance de motifs conditionnels comme des complexes SCF qui détectent des phospho-dégrons ou des motifs créés par des modifications post-traductionnelles, assurant une dégradation strictement régulée,
- la reconnaissance de signaux structuraux internes par des E3 telles qu’APC/C qui reconnaissent des dégrons internes comme le D-box ou la KEN-box, actifs uniquement dans un contexte cellulaire donné,
- la reconnaissance de conformations transitoires ou instables par certaines E3 (CHIP), souvent en coopération avec des chaperonnes, détectent des régions hydrophobes exposées ou des états partiellement dépliés sans que la protéine soit nécessairement " mal repliée " au sens pathologique (
échec du repliement),
- la reconnaissance orientée selon le compartiment comme dans des systèmes spécialisés comme l’ERAD (réticulum endoplasmique) ou la voie MAD (mitochondrie) dans lesquelles les les E3 identifient des protéines mal assemblées, incorrectement insérées ou topologiquement instables dans leur membrane,
- la reconnaissance contextuelle du cycle cellulaire par certaines E3 telles que les complexes SCF ou APC/C, qui ne sont actives qu’à des stades précis du cycle où elles reconnaissent des dégrons conditionnels préalablement activés ou exposés par les complexes cycline/CDK.
Importance biologique
1. Les dégrons constituent le déterminant primaire de la demi-vie d’une protéine.
Ils permettent un contrôle spatio-temporel précis de la protéolyse, essentielle à des processus comme :
- le cycle cellulaire,
- la signalisation,
- la différenciation,
- la réponse au stress.
2. Leur dérégulation est associée à des pathologies majeures :
- cancers, i.e. surexpression de Skp2 ou β-TrCP,
- neurodégénérescences, i.e. mutations de la parkine,
- désordres métaboliques, i.e. défauts d’UBR1/UBR2.
3. Ainsi, le système ubiquitine-protéasome repose sur un dialogue permanent entre signal peptidique (dégron) et enzyme de reconnaissance (E3), garantissant l’équilibre dynamique du protéome cellulaire.


