Modifications post-traductionnelles des protéines
Protéines UBL (Ubiquitin Like)
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

La famille UBL, protéines analogues à l'ubiquitine (UBL pour UBiquitin-Like), remanie ses cibles cellulaires afin de changer leurs fonctions.
Le système ubiquitine-protéasome (UPS) et le système ubiquitine-like (UBL) suivent une voie analogue (Origin and Function of Ubiquitin-like Protein Conjugation 2009 et Structure and evolution of ubiquitin and ubiquitin-related domains 2012).
Vue d'ensemble des UBL
1. Une famille croissante de protéines analogues à l'ubiquitine (UBL pour UBiquitin-Like) modifie aussi leurs cibles cellulaires (Protéasome, ubiquitine et protéines apparentées à l'ubiquitine 2011).
(Figure : vetopsy.fr d'après Geng)
Les UBL régulent un grand nombre de processus cellulaires :
- le transport nucléaire,
- la protéolyse,
- la traduction,
- l'autophagie
- les processus antiviraux (Ubiquitin-Like Proteins 2012).
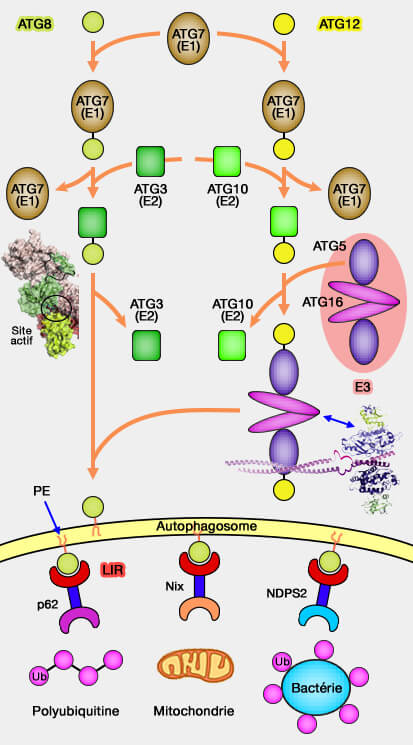
2. Les protéines UBL, outre leur structure tertiaire similaire à l'ubiquitine, i.e. β-GRASP fold, peuvent être conjuguées sur des résidus lysines de substrats, en faisant intervenir des processus enzymatiques identiques.
- L'enzyme d'activation E1 active l'UBL sous la dépendance de l'ATP,.
- L'enzyme de conjugaison E2 la transfère sur le substrat.
- L'E3 ligase, généralement, mais qui peut être absente, facilite ce transfert.
Certaines UBL sont transférées non à une protéine, mais à un lipide, comme ATG8 par exemple.
Membres des UBL
Les Ubl (Ubiquitin-Like protein) comprennent, pour l'instant, une dizaine de membres, dont l'ubiquitine (Origin and Function of Ubiquitin-like Protein Conjugation 2010).
(Figure : vetopsy.fr d'après Ronau)
Protéines jouant différents rôles
Des UBL jouent des rôles variés.
1. les SUMO.
2. NEDD8(Neural precursor cell Expressed, Developmentally Down-regulated 8) est un interrupteur moléculaire des ligases Cullin-RING (CRL).
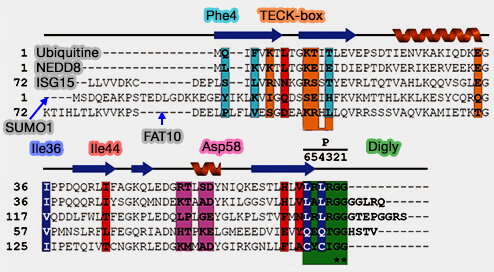
3. ISG15 (Interferon-Stimulated Gene 15) agit dans l'ISGylation, i.e. addition covalente d'ISG15 aux protéines membranaires et cytoplasmiques.
- UBE1L, l'enzyme d'activation E1, forme une liaison thioester ($\ce{R-S-CO-R'$}), liaison " riche en énergie " avec ISG15 et le transfert à UbcH8, l'enzyme de conjugaison E2 (The UbcH8 ubiquitin E2 enzyme is also the E2 enzyme for ISG15, an IFN-α/β-induced ubiquitin-like protein 2004).
- UbcH8 transfère enfin ISG15 à HERC5, E3 ligase de la famille des HECT.
L'UBP43, aussi connu sous USP18, de la famille des DUB, est la seule protéase connue pour la deISGylation.
4. FAT10 agit dans les tissus du système immunitaire (FAT10, a Ubiquitin-Independent Signal for Proteasomal Degradation 2005 et FAT10, an ubiquitin-like protein, confers malignant properties in non-tumorigenic and tumorigenic cells 2014)
(Figure : vetopsy.fr d'après Huxley et van der Veen)
5. URM1 modifie les ARN de transfert, ce qui reflète les origines du système des UBL dans l'évolution par le transfert du soufre dans la biosynthèse de la thiamine et la molybdoptérine chez les bactéries qui utilisent le β-GRASP fold (Biosynthesis and functions of sulfur modifications in tRNA 2014).
6. HUB1 (Homologous to Ub, aussi appelé Ubl5 ou beacon chez les Mammifères) ;
7. UFM1 (Ubiquitin-Fold Modifier 1) intervient dans le stress cellulaire au niveau du réticulum endoplasmique (Mechanistic Study of Uba5 Enzyme and the Ufm1 Conjugation Pathway 2014).

Vous pouvez consulter une figure qui résume les mécanismes.
Protéines et
autophagie
On trouve aussi des protéines qui jouent un rôle dans l'autophagie (Ubiquitination and selective autophagy 2013 et Atomistic Autophagy: The Structures of Cellular Self-Digestion 2014).

L'autophagie est étudiée dans de nombreux chapitres spécifiques.
1. Les macromolécules, les organites comme les mitochondries et les bactéries pathogènes doivent être " sélectionnés " pour être séquestrés dans un compartiment à double membrane, i.e. l'autophagosome, puis hydrolysés dans les lysosomes.
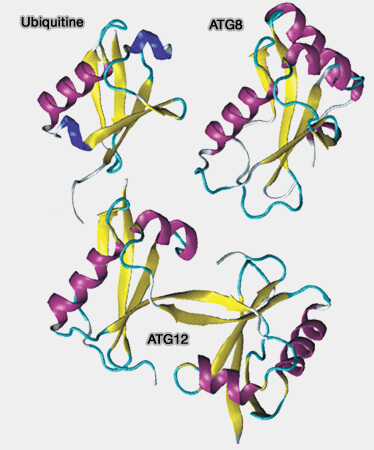
2. ATG8 et ATG12, deux protéines faisant partie des ATG (AuTophaGy-related protein), régulent la formation des autophagosome (
Structure des UBL
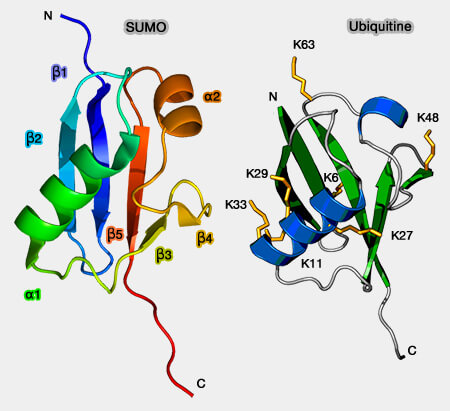
1. Les UBL possèdent un domaine UBL (Ubiquitine-Like domain), i.e. même repli, le β-GRASP fold, que celui de l'ubiquitine, mais une structure primaire différente :
- Ce feuillet-β à 5 brins et deux hélices-α se retrouve dans l'ubiquitine.
- Par contre, les UBL ne possèdent pas les résidus glycine C-terminaux requis pour la conjugaison à la lysine cible du substrat (Encyclopedia of cell biology 2015).
(Figure : vetopsy.fr)
Certains domaines sont appelés UFD (ubiquitin-fold domains), comme par exemple dans SAE1 (SUMO-activating enzyme E1) ou l'enzyme d'activation E1 (E1 ubiquitin-activating enzyme) de l'ubiquitine.
2. Ce domaine UBL se retrouve dans les désubiquitinases (DUB), en particulier dans les USP, et joue des rôles très différents dans la régulation de leur activité (rôle des domaines UBL dans les DUB).
a. Certaines protéines à UBD contiennent aussi un domaine UBL, domaine qui peut interagir avec des domaines UBD :
- soit au sein de la même protéine,
- soit entre deux protéines distinctes.
b. Ces protéines peuvent adresser des substrats ubiquitinés au protéasome 26S, en interagissant avec les substrats ubiquitinés via leurs domaines UBD et avec le protéasome via leurs domaines UBL, comme dans le cas de Rad23, DSK2 ou Ddi1.
On les retrouve aussi dans d'autres protéines comme les immunoglobulines, mais aussi chez les protéines à domaine DIX comme Dischvelled, - l'hélice α2 entre β4 et β5 est absente -, l'axine ou Ccd1/DIXDC1.
3. Le résultat des modifications de ces UBL est que l'on retrouve une topologie similaire :
- soit à celle de l'ubiquitine, i.e. même taille et même forme, comme pour SUMO, NEDD8 et ATG12,
- soit à celle d'un dimère comme ISG15 ou FAT10.
(Figure : vetopsy.fr d'après Kish-Trier)
a. Les désubiquitinases (DUB) savent faire la différence entre UBL et ubiquitine par, entre autres, les 4 résidus qui précèdent le motif Gly-Gly terminal de l'ubiquitine (Leu71-Arg72-Leu73-Arg74-Gly75-Gly76).
- ISG15 possède un motif identique et NEDD8 un motif très proche (Leu-Ala-Leu-Arg-Gly-Gly) sont hydrolysés par des DUB identiques à celles de l'ubiquitine.
- Toutefois, les E1 différencient NEDD8 de l'ubiquitine par son Ala72.
- Ceux de SUMO (Gln-Gln/Glu-Gln-Thr-Gly-Gly), ATG12 (Lys-Ser-Gln-Ala-Trp-Gly) et FAT10 (Ser-Tyr-Cys-Ile-Gly-Gly) sont différents.
b. Les UBL sont reconnus, au moins pour SUMO, par un motif SIM/SBD (SUMO-Interaction/Binding Motif).


