Modifications post-traductionnelles des protéines
Ubiquitination : E3 ubiquitine ligases de la famille RING
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

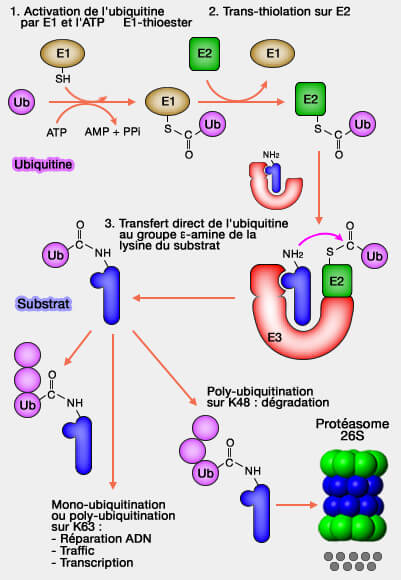
Les ligases E3 de la famille RING catalysent le transfert direct de l’ubiquitine du complexe E2~Ub vers le substrat, assurant une ubiquitination rapide et hautement sélective.
Les E3 ubiquitine ligases sont classées en trois grandes familles (New insights into ubiquitin E3 ligase mechanism 2014 et HECT and RING finger families of E3 ubiquitin ligases at a glance 2012).
- la famille HECT,
- la famille RING, à domaine RING ou RING-related, les U-box ne contenant pas d'atomes de zinc y sont intégrées,
- la famille RBR (RING-Between-RING).
(Figure : vetopsy.fr)
Vue d'ensemble
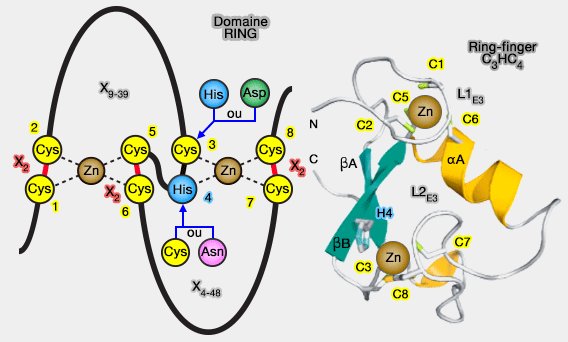
1. La famille RING (Really Interesting New Gene) regroupe plus de 600 ligases E3 chez les mammifères.
a. Elle se distingue par un domaine RING, coordonnant deux ions Zn++, essentiel à la stabilisation de la structure et à l’interaction avec l’enzyme E2.
b. Toutefois, on y ajoute des protéines qui possèdent :
- des domaines RING-related, comme le domaine PHD ou LAP,
- les U-box dont le domaine, de structure tertiaire identique au domaine RING, ne contient pas d'atomes de zinc.
(Figure : vetopsy.fr d'après Berndsen)
2. Contrairement aux ligases de la famile HECT ou RBR, les ligases RING catalysent le transfert direct de l’ubiquitine du complexe E2~Ub vers la lysine du substrat, sans formation de liaison intermédiaire avec l’E3 (
a. Dans certaines ligases, souvent sous forme dimérique, le domaine de reconnaissance du substrat et le domaine RING, ou équivalent comme les U-box, sont situés sur la même protéine.
b. Dans d'autres, les deux domaines sont situés sur des protéines différentes.
c. Les E3 reconnaissent :
- le domaine UBC des E2, domaine de conjugaison à l'ubiquitine qui contient la cystéine catalytique,
les extensions N- et C-terminales des E2 contribuent à leur spécialisation et à la sélectivité de l’interaction avec la ligase RING.
Organisation structurelle des ligases RING
Les ligases RING se répartissent en deux grandes catégories selon leur organisation (RING Domain E3 Ubiquitin Ligases 2009 et RING-type E3 ligases: Master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination 2014) :
- les RING monomériques et dimériques,
- les complexes RING.
RING monomériques et dimériques
1. Les RING monomériques et dimériques :
- ne comportent aucun adaptateur,
- intègrent dans la même entité protéique, ou dans un dimère fonctionnel, le domaine RING et le site de reconnaissance du substrat,
- assurent une ubiquitination directe, sans charpente de type culline.

Les sites de reconnaissance des dégrons et des substrats par les ligases E3 sont étudiés dans un chapitre spécifique.
2. Ces RING peuvent fonctionner sous forme :
(Figure : vetopsy.fr)
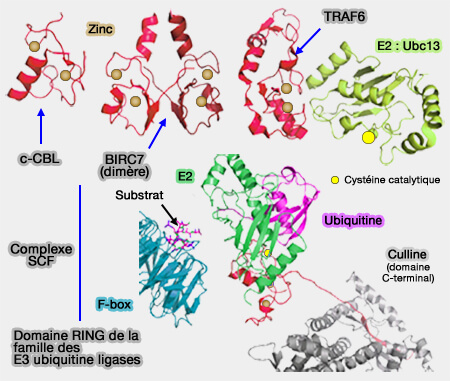
a. de monomères comme c-CBL- Casitas B-lineage Lymphoma - (Structure of a c-Cbl-UbcH7 Complex: RING Domain Function in Ubiquitin-Protein Ligases 2000).
b. d'homodimères comme :
- BIRC7 (Baculoviral IAP Repeat-Containing protein 7) qui comprend deux hélices α qui contribuent aux contacts (Structure of BIRC7-E2 ubiquitin conjugate reveals the mechanism of ubiquitin transfer by a RING dimer 2012),
- CHIP et Prp19 qui sont des U-box,
- TRAF6 (Tumor necrosis factor Receptor Associated Factor 6) qui s'enroule autour de l'enzyme de conjugaison E2 (E2 interaction and dimerization in the crystal structure of TRAF6 2009),
- Ubc13…
RING multiprotéiques
Les RING multiprotéiques regroupent les ligases dont le module catalytique RING est intégré dans un assemblage de grande taille, généralement organisé autour d’une sous-unité échafaudage et d’un ensemble de sous-unités régulatrices ou adaptatrices.
1. Ils comprennent notamment :
- les complexes CRL (Cullin-RING ligases), organisés autour d’une culline canonique associée à un module RING,
- le complexe APC/C, dont le module catalytique APC2-APC11 adopte une architecture apparentée, bien que fonctionnellement distincte.
2. Ces complexes permettent une spécificité étendue, une régulation hiérarchisée et un contrôle fin de l’ubiquitination de nombreux substrats.
Familles des E3 ubiquitine ligases RING
Ligases Cullin-RING (CRL)
Les ligases Cullin-RING (CRL) représentent la plus vaste sous-famille des E3 ubiquitine ligases de type RING.

Vu leur importance, les ligases Cullin-RING (CRL) sont étudiées dans un chapitre spécifique.
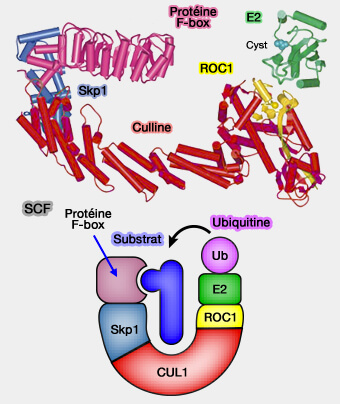
1. Les CRL reposent sur une charpente modulaire en culline, associée à :
- une sous-unité RING (Rbx1/Roc1) qui recrute l’enzyme de conjugaison E2,
- des adaptateurs variables, i.e. Skp1, élongines, BTB, DDB1… assurant la reconnaissance du substrat.
(Figure : vetopsy.fr)
a. Ce système modulaire permet de constituer de multiples complexes CRL parmi lesquels le complexe SCF (Skp1-Cullin1-F-box), pivot de la dégradation régulée des protéines du cycle cellulaire.
b. Ces ligases orchestrent la majorité des ubiquitinations conduisant à la dégradation protéasomale, tout en intégrant de multiples mécanismes de régulation (
- neddylation des cullines, activant le complexe,
- inhibition transitoire par CAND1, empêchant l’assemblage prématuré,
- remplacement dynamique des adaptateurs, modulant la spécificité du substrat.
2. Le complexe APC/C (Anaphase-Promoting Complex/Cyclosome), constitue une ligase RING multiprotéique " CRL-like ".
a. Son module catalytique repose sur :
- APC2, une protéine analogue de la culline (cullin-like), jouant un rôle d’échafaudage, mais non neddylée,
- APC11, une sous-unité RING assurant le transfert direct de l’ubiquitine depuis l’E2~Ub.
b. Bien qu’il partage une architecture structuralement proche des CRL, l’APC/C n’est pas une CRL au sens strict, car il n’utilise aucune culline canonique et ne dépend pas du cycle CSN-CAND1-NEDD8.
c. L’ensemble contrôle la transition métaphase-anaphase et la dégradation cyclique des cyclines au cours de la mitose.
Famille des RING-related
Ces E3 ubiquitine ligases comportent des domaines apparentés au domaine RING, partageant une architecture à doigt de zinc, mais une topologie variable qui comprennent notamment :
- les domaines PHD finger (Plant HomeoDomain) qui ressemblent aussi aux domaines FYVE,
- les domaines LAP (Leukemia Associated Protein), riches en cystéines (The leukemia-associated-protein (LAP) domain, a cysteine-rich motif, is present in a wide range of proteins, including MLL, AF10, and MLLT6 proteins 1995).
Famille des U-Box
Les ligases U-box possèdent un domaine U-box proche du domaine RING, mais sans atome de zinc.
Elles fonctionnent souvent sous forme d'homodimères et d'hétérodimères spécifiques par l'interaction entre leurs domaines RING/RING, RING/U-box et U-box/U-box
Ligases RING particulières de type UBR
Certaines ligases RING appartiennent à un groupe fonctionnel particulier appelé UBR (Ubiquitin-protein ligases recognizing N-degrons), ou N-recognins, spécialisées dans la reconnaissance des acides aminés N-terminaux déstabilisants selon la voie de la règle du N-terminal (N-end rule).
1. Elles combinent :
- un domaine UBR-box, chargé de la reconnaissance,
- un domaine RING catalytique assurant le transfert d’ubiquitine depuis l’enzyme E2.
2. Les principales ligases de ce groupe sont UBR1, UBR2 et UBR4, qui appartiennent structuralement à la famille RING.
Elles interviennent dans la surveillance de la qualité des protéines et le contrôle de leur stabilité en identifiant des signaux internes ou terminaux.
3. À l’inverse, UBR5 (EDD1), bien que fonctionnellement apparentée à ce groupe, contient un domaine HECT actif et relève des HECT atypiques, dépourvues de domaines RLD et WW.
Ces ligases illustrent la plasticité fonctionnelle du système ubiquitine dans lequel certaines E3 RING combinent une reconnaissance directe du substrat avec une activité catalytique indépendante d’adaptateurs, marquant ainsi une zone de transition entre les familles RING et HECT.


