Modifications post-traductionnelles des protéines
Ubiquitination : rôles
- Biochimie
- Chimie organique
- Bioénergétique
- Composition de la matière vivante
- Composés organiques
- Protides
- Acides nucléiques
- Glucides
- Lipides
- Hormones
- Composés inorganiques
- Composés organiques
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

L'ubiquitination régule la stabilité, la localisation et les interactions des protéines, contrôlant ainsi la signalisation, le trafic membranaire et la dynamique nucléaire.
Son rôle et sa régulation dépendent du type d’attachement de l’ubiquitine, du contexte cellulaire et du substrat concerné.
Vue d'ensemble des rôles de l'ubiquitination
Les protéines ubiquitinées voient leurs propriétés de liaison, de stabilité, de localisation ou d’activité profondément modifiées avec les autres protéines, ou parfois, avec les lipides (Catalytic Mechanism and Assembly of the Proteasome 2009).
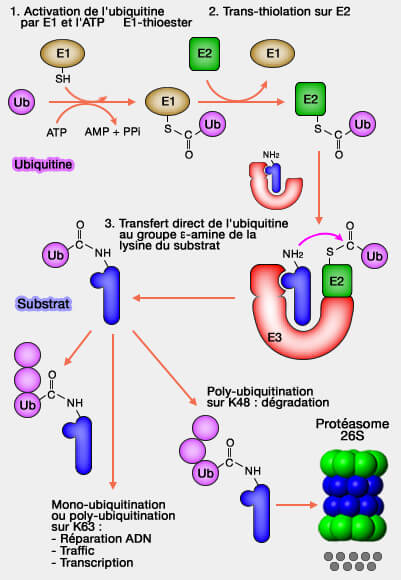
(Figure : vetopsy.fr)
1. L’identification systématique des sites d’ubiquitination, grâce à des peptides marqués par un α-diGly a permis de répertorier près de 19 000 sites d’ubiquitination sur environ 5 000 protéines (Systematic and Quantitative Assessment of the Ubiquitin-Modified Proteome 2011).
Cette densité souligne l’omniprésence du marquage ubiquitine dans la régulation cellulaire.
2. De plus, les E3 ubiquitine ligases contiennent de nombreux motifs qui interagissent avec d'autres protéines sans pour autant les dégrader, par exemple :
- à certains moments du cycle cellulaire, du développement ou de la différenciation cellulaire,
- en réponse à certains stimuli comme lors de la signalisation cellulaire.
2. La régulation temporelle et contextuelle de l’ubiquitination dépend (
- du niveau d’expression des ligases E3 ou de leurs substrats,
- de cofacteurs régulateurs, lors de l'activation des Smurfs par Smad7 (I-Smad) ou de la neddylation par NEDD8 des cullines,
- des phosphorylations préalables des substrats, nécessaires à leur reconnaissance par certaines E3, comme les F-BOX des SCF…

Le rôle et la régulation de l'ubiquitination sont spécifiques à chaque protéine.
Dégradation de la protéine ubiquitinée
1. La monoubiquitination ou la polyubiquitination doit être effectuée sur des lysines particulières, i.e. K48 et K29 par exemple, pour la présentation de la protéine au protéasome 26S pour être détruite (" baiser moléculaire de la mort ").
Un exemple classique est la dégradation de p53 après polyubiquitination par la ligase MDM2.
2. Les protéines du réticulum endoplasmique, sont également ciblées par un mécanisme analogue, le système ERAD (Endoplasmic Reticulum-Associated Degradation), assurant la rétrotranslocation des protéines mal conformées vers le cytosol avant leur destruction
Remarque : un processus apparenté est la sumoylation, qui module la stabilité de nombreux régulateurs nucléaires.
Fonctions non protéolytiques
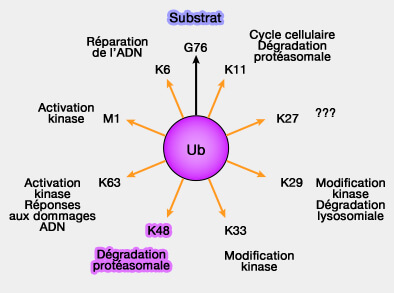
(Figure : vetopsy.fr)
Des polyubiquitinations ou des monoubiquitinations remplissent des fonctions non dégradatives, en modulant les interactions, la localisation ou les réponses signalétiques.

Vous pouvez lire : Identification des " Ubiquitin Specic proteases " impliquées dans la régulation des voies de l'immunité chez la drosophile (2009).
Interactions
protéine/protéine
1. Les domaines UBD (Ubiquitin Binding Domain) régulent la reconnaissance des protéines mono- ou polyubiquitinées, en particulier dans la signalisation cellulaire.
On en compte plus de 20 différentes familles et leur nombre ne cesse d'augmenter.
- La classe la plus nombreuse est celle des UBD de structure α-hélicale qui se lient au patch hydrophobe Ile4.
- La deuxième classe est celle des zinc-finger (ZNF) qui reconnaissent trois régions différentes sur la surface de l'ubiquitine.
2. La conjugaison et la déconjugaison de l'ubiquitine et des UBL (ubiquitin-like protein) aux protéines cellulaires sont des processus régulés de l'homéostasie cellulaire (Substrate specificity of the ubiquitin and Ubl proteases 2016).
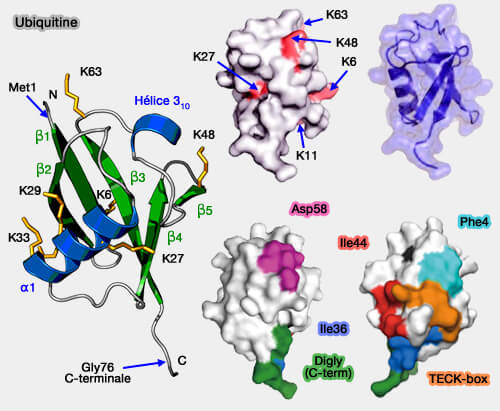
(Figure : vetopsy.fr d'après Hurley et Ronau)
3. Les interactions entre protéines ubiquitinées et UBD déterminent la spécificité fonctionnelle des signaux ubiquitine.
Elles reposent sur des liaisons non covalentes entre les UBD et les surfaces hydrophobes ou électrostatiques de l’ubiquitine.
a. Elles sont dépendantes de la mono- ou de la polyubiquitination, et dans ce cas de la conformation de la chaîne d'ubiquitine.
- Les chaînes K48 adoptent une structure compacte.
- Les chaînes K63 présentent une organisation linéaire et étendue, modulant la reconnaissance des UBD.
b.Les interactions sont complexes et dépendent de nombreux facteurs :
- multiplication des sites d’ubiquitination dans une même protéine,
- oligomérisation des protéines contenant des UBD, augmentant l’avidité et la sélectivité,
- diversité des contextes cellulaires, i.e. complexes de signalisation, endosomes, noyau, cytosquelette.
Endocytose
1. L'endocytose internalise et trie des récepteurs membranaires par leur ubiquitination (Ubiquitin-dependent endocytosis, trafficking and turnover of neuronal membrane proteins 2014 et Proteasome-Independent Functions of Ubiquitin in Endocytosis and Signaling 2009).
a. De nombreux récepteurs, comme EGFR, sont ubiquitinés pour être internalisés puis dirigés vers les lysosomes pour dégradation (Membrane Protein Targeting to the MVB/Lysosome 2009),
b. D’autres, au contraire, sont recyclés vers la membrane plasmique, comme certains transporteurs neuronaux, selon un contrôle dépendant :
- les DUB (désubiquitinases).,
- des régulateurs comme Rabex-5 (Molecular mechanism for Rabex-5 GEF activation by Rabaptin-5 2014).
Elle peut les recycler comme pour (Ubiquitin-dependent endocytosis, trafficking and turnover of neuronal membrane proteins 2014 et Proteasome-Independent Functions of Ubiquitin in Endocytosis and Signaling 2009) :
- Rabex-5 (Molecular mechanism for Rabex-5 GEF activation by Rabaptin-5 2014),
- les DUB (désubiquitinases).
2. Les régulateurs de l'endocytose, qui possèdent le plus souvent des UBD, trient aussi les cargos pour leur adressage aux lysosomes.
Rôles sur l'ADN

(Figure : vetopsy.fr d'après Dupond sous Open Archive)
1. La transduction de l'ADN dans les cellules embryonnaires et la différenciation des cellules dépendent de la compaction des nucléosomes et des modifications chimiques des histones (
- Ces modifications incluent la méthylation, l’acétylation, la phosphorylation et l’ubiquitination (Regulation of Receptors and Transporters by Ubiquitination: New Insights into Surprisingly Similar Mechanisms 2007).
- L'ubiquitination, comme celle des Smad4 par l'Ectodermine/TRIM33, ne provoque pas la destruction de la protéine, mais régule son incorporation dans des complexes protéiques.
2. La réparation de l'ADN implique l'ubiquitination du facteur de réplication de l’ADN (PCNA - Proliferating Cell Nuclear Antigen -), monoubiquityné sur Lys164 pour recruter des polymérases translesionnelles lors de la tolérance aux dommages de l’ADN.
PCNA peut également être sumoylé pendant la phase S du cycle cellulaire, favorisant la fidélité de la réplication en prévenant le recours inapproprié à la recombinaison homologue.
Autres rôles de l'ubiquitination
L’ubiquitination intervient également dans plusieurs processus spécialisés, au-delà de la dégradation, de la signalisation et de la régulation nucléaire.
1. Elle participe à la mitophagie sélective, où la ligase parkine, activée par PINK1, marque les protéines de la membrane mitochondriale pour leur élimination via l’autophagie.
Ce mécanisme assure le recyclage des mitochondries endommagées et la prévention du stress oxydatif. Elle contribue à la réponse antivirale innée.
2. La polyubiquitination K63 de MAVS ou de TRAF3 active la kinase TBK1 et favorise la production d’interférons de type I, essentielle à la défense cellulaire contre les virus.
3. L’ubiquitination des ribosomes participe au contrôle qualité et à la régulation du cycle cellulaire (Cell cycle-regulated modification of the ribosome by a variant multiubiquitin chain 2000).
La ligase Listerine (Ltn1) ubiquitine les chaînes polypeptidiques bloquées sur les ribosomes, déclenchant leur dégradation par le protéasome et maintenant l’intégrité du système de traduction.
4. Enfin, l’ubiquitination régule la dynamique des complexes multiprotéiques, en facilitant l’assemblage ou le démantèlement de structures telles que les inflammasomes ou les complexes TNFR1, assurant un ajustement rapide des voies de signalisation.
Perspectives et implications pathologiques
1. Les dérégulations du système ubiquitine-protéasome (UPS) sont impliquées dans de nombreuses pathologies humaines :
- la prolifération cellulaire et l'oncogenèse, par déséquilibre entre ubiquitination et désubiquitination,
- la neurodégénérescence par accumulation de protéines mal éliminées,
- la réponse inflammatoire excessive ou défectueuse.
2. On peut citer plusieurs exemples.
- UBA1, enzyme d’activation E1, est au cœur du syndrome VEXAS, dans lequel des mutations somatiques altèrent l’amorçage du cycle ubiquitine et déclenchent une inflammation systémique associée à une cytopénie (Ubiquitin-Activating Enzyme E1 (UBA1) as a Prognostic Biomarker and Therapeutic Target in Breast Cancer: Insights into Immune Infiltration and Functional Implications 2024).
- La parkine, E3 de la famille RBR, voit sa perte de fonction conduire à une défaillance du recyclage protéique, à l’accumulation de substrats toxiques et à la neurodégénérescence, mécanismes centraux dans la maladie de Parkinson (Structure of Parkin Reveals Mechanisms for Ubiquitin Ligase Activation 2013).
- NEDD4, ITCH et HUWE1, E3 de type HECT, contribuent à la tumorigenèse et à la résistance à l’apoptose lorsque leur activité est dérégulée, soulignant leur rôle critique dans la transformation cellulaire ( (HECT E3 ubiquitin ligases - emerging insights into their biological roles and disease relevance 2020).


