Système endo-lysosomal
Complexe ESCRT
Vps4 et recyclage de ESCRT-III
- Vue d'ensemble du système endomembranaire
- Endosomes
- Vue d'ensemble des endosomes
- Endosomes précoces
- Endosomes de recyclage
- Vésicules intraluminales (ILV) et endosomes ou corps mutivésiculaires (MVE/MVB)
- Endosomes tardifs
- Système endo-lysosomal et phosphoinositides
- MCS (sites de contact membranaire) RE/endosomes/lysosomes
- Lysosomes
- Trafic des endosomes/lysosomes
- Fusion et fission membranaire
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

L'extrémité C-terminale de ESCRT-III recrute plusieurs molécules effectrices dont la protéine VPS4 (AAA ATPase vacuolaire de tri 4) qui a un rôle essentiel dans le désassemblage et le recyclage de l'ESCRT-III.
Le complexe ESCRT (Endosomal Sorting Complex Required for Transport ou complexe de tri endosomal requis pour le transport) est composé de 5 complexes :
- ESCRT-0,
- ESCRT-I,
- ESCRT-II,
- ESCRT-III,
- Vps4 (Vacuolar Protein Sorting 4) qui contient un domaine catalytique AAA+ ATPase.
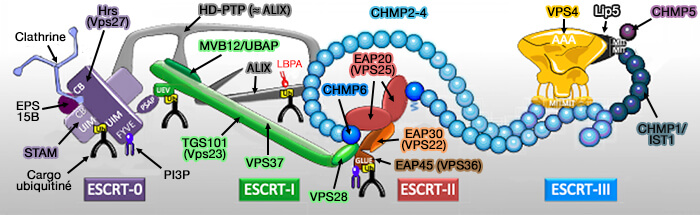
(Figure : vetopsy.fr d'après Christ et Hill)
L'extrémité C-terminale de ESCRT-III recrute plusieurs molécules effectrices dont la protéine VPS4 (AAA ATPase vacuolaire de tri 4) qui a un rôle essentiel dans : :
- le désassemblage et le recyclage de l'ESCRT-III, i.e. formes solubles des CHMP,
- le remodelage de la membrane et la scission de la vésicule (The ESCRT machinery: From the plasma membrane to endosomes and back again 2015).
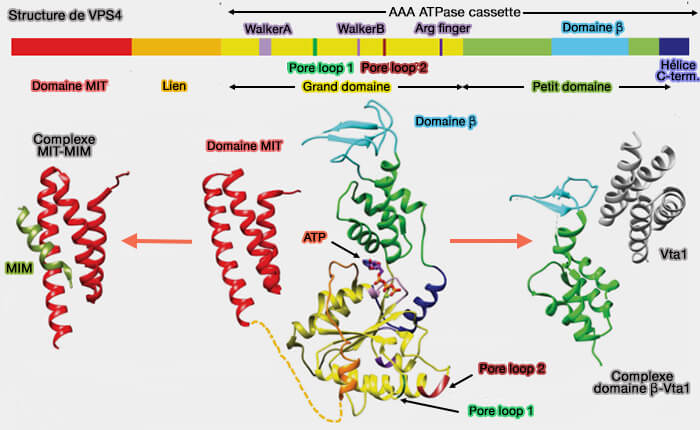
Structure de Vps4
1. VPS4 fait partie de la grande famille des AAA+ ATPases qui a été classée en plusieurs clades, dont le clade méiotique avec les enzymes spastine, katanine et fidgétine.
Toutes ces enzymes désassemblent des structures protéiques polymères spécifiques :
- Vps4, les polymères ESCRT-III,
- la spastine, la katanine p60 et la fidgétine affectant de multiples aspects de la dynamique cellulaire en sectionnant les microtubules (
rôles de la spastine avec ESCRT-III dans les endosomes de recyclage).
(Figure : vetopsy.fr d'après Han et Hill)
2. La structure de VPS4 comprend (Structure and mechanism of the ESCRT pathway AAA+ ATPase Vps4 2019) :
- un domaine MIT (Microtubule Interaction and Transport) N-terminal,qui se lie aux domaines MIM des sous-unités ESCRT-III,
- un domaine catalytique AAA+ ATPase, relié à MIT par un lien flexible (Meiotic Clade AAA ATPases: Protein Polymer Disassembly Machines 2016).
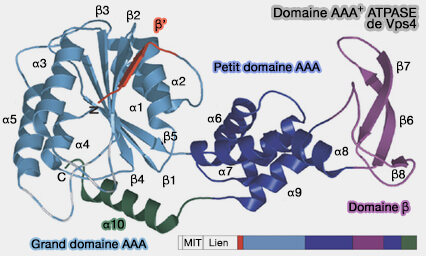
Domaine AAA+ ATPase de Vps4
(Figure : vetopsy.fr d'après Scott
et coll)
3. La cassette ATPase comprend un grand et un petit domaine ATPase.
- Le grand domaine ATPase contient les motifs Walker A et Walker B qui conduisent respectivement à la liaison et à l'hydrolyse de l'ATP et à la formation des boucles de pores qui engagent le substrat dans le pore central de l'hexamère (Structural and mechanistic studies of VPS4 proteins 2005).
- Le petit domaine ATPase comprend quatre hélices, suivies d'une hélice C-terminale qui le compacte contre le grand domaine.
Le domaine β émane d'une boucle interne du petit domaine ATPase et interagit avec LIP5/Vta1.
Héxamérisation de Vps4
1. VPS4, comme de nombreuses autres AAA+ ATPases est activée sous forme hexamérique avec la présence d'un pore central (The oligomeric state of the active Vps4 AAA ATPase 2014).
VPS4 utilise l'énergie de l'hydrolyse de l'ATP pour activer la translocation du cargo dans le pore central de l'hexamère et le déplier (Mechanistic insights into bacterial AAA+ proteases and protein-remodelling machines 2016).
a. De nombreuses structures de Vps4 ont été révélées, dont certaines sont trompeuses, et il semble qu'à l'heure actuelle, un consensus s'effectue sur une conformation hexamérique asymétrique ( Structural basis of protein translocation by the Vps4-Vta1 AAA ATPase 2017 et Mechanism of Vps4 hexamer function revealed by cryo-EM 2017 et Cryo-EM structures of the ATP-bound Vps4E233Q hexamer and its complex with Vta1 at near-atomic resolution 2017).
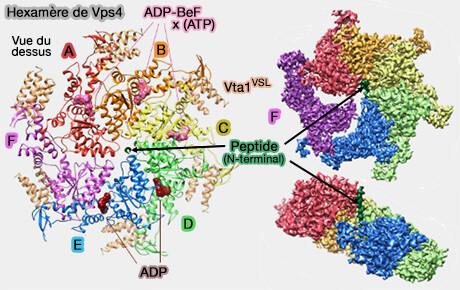
(Figure : vetopsy.fr d'après Han et coll)
b. En dépit de la similitude des trois structures récemment signalées, l'interprétation du mécanisme est différente.

Pour une revue, vous pouvez lire : Structure and mechanism of the ESCRT pathway AAA+ ATPase Vps4 (2019).
2. L'hexamère Vps4 comprendrait dans ce modèle hexamérique asymétrique (The AAA ATPase Vps4 binds ESCRT-III substrates through a repeating array of dipeptide-binding pockets 2017 avec de nombreuses vidéos explicatives) :
- un ensemble hélicoïdal de cinq sous-unités (sous-unités A-E),
- une sixième sous-unité (F) qui se situe à l'extrémité de la sous-unité E (
héxamérisation asymétrique).
2. L'hélice à cinq sous-unités est droite avec une translation de ~ 6,3Å et une rotation de 60° entre les sous-unités, i.e. correspondant à la symétrie du peptide ESCRT-III lié, qui adopte une conformation de feuillet β (β-strand).
- Ces spirales autour de l'axe hélicoïdal se lient dans une rainure formée par les cinq sous-unités hélicoïdales VPS4. Les peptides de substrat successifs, DEIVNKVL dans l'article, se lient de la même manière aux interfaces successives entre les sous-unités VPS4.
- La chaîne latérale du premier résidu de chaque dipeptide se lie dans une poche de classe I, tandis que la chaîne latérale du deuxième résidu de chaque dipeptide se lie dans une poche de classe II.
- Ces poches sont répétées à chacune des quatre interfaces des sous-unités VPS4 hélicoïdales.
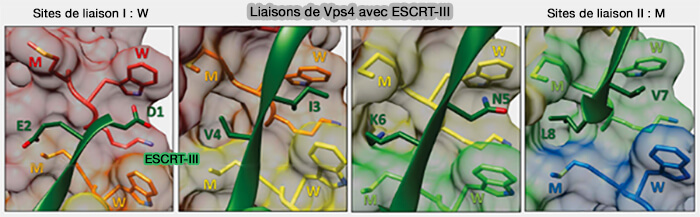
(Figure : vetopsy.fr d'après Han et Hill)
3. Les sites de liaison :
- de classe I sont principalement formées par le résidu de tryptophane (W) de la boucle de pore 1 qui forme une encoche hydrophobe qui apparaît capable d'accueillir une grande variété de chaînes latérales d'acides aminés (premier résidu) entre deux sous-unités VPS4 adjacentes,
- de classe II contiennent une méthionine (M) hydrophobe flanquée par la boucle de pore 2 qui peut servir de poche, avec la sous-unité adjacente, à une grande variété d'acides aminés.

La liaison est largement indépendante de la séquence pour la translocation du substrat de protéines ESCRT-III.
Recyclage de ESCRT-III
Interactions entre Vps4 et ESCRT-III
Interactions MIT/MIM des CHMP

Le recyclage induit par Vps4 dépend de l'interaction du domaine MIT (Microtubule Interacting and Trafficking molecule domain) avec les motifs d'interaction MIM (Mit Interaction Motif) des sous-unités ESCRT-III.
(Figure : vetopsy.fr d'après Huxley et Yang)
Les domaines MIT se composent de trois faisceaux d'hélices qui interagissent avec des partenaires protéiques via plusieurs interfaces.
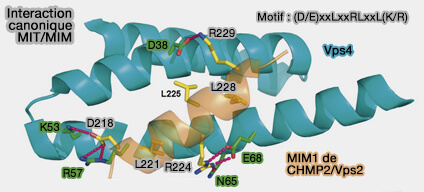
Interaction canonique MIT/MIM
(Figure : vetopsy.fr d'après Hurley et Yang)
1. L'interaction canonique d'ESCRT-III avec le domaine MIT de Vps4 se produit à travers MIM1 des sous-unités :
- CHMP2B/Vps2 (Structural basis for selective recognition of ESCRT-III by the AAA ATPase Vps4 2007),
- CHMP1A/Did2 (ESCRT-III recognition by VPS4 ATPases 2007)
- IST1 (Distinct Mechanisms of Recognizing Endosomal Sorting Complex Required for Transport III (ESCRT-III) Protein IST1 by Different Microtubule Interacting and Trafficking (MIT) Domains 2015)
Le MIM1 consiste en une seule hélice qui interagit avec les hélices α2 et 3 du domaine MIT de Vps4 via plusieurs résidus Leu clés de MIM1 (D/E)xxLxxRLxxL(K/R).
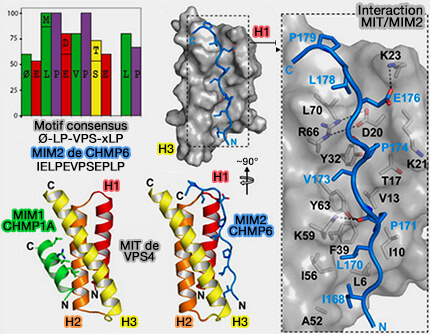
MIT de Vps4 avec MIM2 de CHMP6
(Figure : vetopsy.fr d'après Kieffer
et coll)
2. Une autre interaction s'effectue entre la conformation étendue MIM2 de la sous-unité ESCRT-III de CHMP6 et les hélices α1 et α3 du domaine MIT de VPS4 (Two Distinct Modes of ESCRT-III Recognition Are Required for VPS4 Functions in Lysosomal Protein Targeting and HIV-1 Budding 2008).
CHMP4 et CHMP5 interagissent également avec Vps4-MIT via des motifs MIM2, mais très faiblement par rapport à CHMP6 (Structural Basis of Molecular Recognition between ESCRT-III-like Protein Vps60 and AAA-ATPase Regulator Vta1 in the Multivesicular Body Pathway 2012).
Interactions des CHMP
sans MIM
CHMP3 n'a pas de MIM interagissant avec VPS4, mais CHMP3 et la plupart des autres sous-unités ESCRT-III ont des séquences qui se lient à d'autres protéines à domaines MIT.
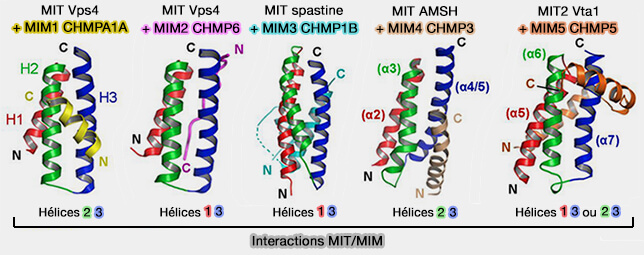
(Figure : vetopsy.fr d'après Yang et coll)
1. LIP5/Vta1 est un cofacteur de VPS4 (Novel Interactions of ESCRT-III with LIP5 and VPS4 and their Implications for ESCRT-III Disassembly 2008 et Structural Basis of Vta1 Function in the Multivesicular Body Sorting Pathway 2008).
- Le domaine β est un insert du petit domaine AAA qui interagit avec le domaine VSL (Vta1/SBP-1/Lip5) C-terminal de LIP5 et permet son oligomérisation essentielle à sa fonction (Vps4 Stimulatory Element of the Cofactor Vta1 Contacts the ATPase Vps4 α7 and α9 to Stimulate ATP Hydrolysis 2014).
- En outre, LIP5 contient une paire de domaines MIT (MIT1 et MIT2) en tandem à son extrémité N-terminale qui se lient directement aux sous-unités ESCRT-III, i.e. augmentant ainsi les signaux d'activation de ESCRT-III à VPS4 (ESCRT-III Family Members Stimulate Vps4 ATPase Activity Directly or via Vta1 2008 et Structural Basis of Molecular Recognition between ESCRT-III-like Protein Vps60 and AAA-ATPase Regulator Vta1 in the Multivesicular Body Pathway 2012).
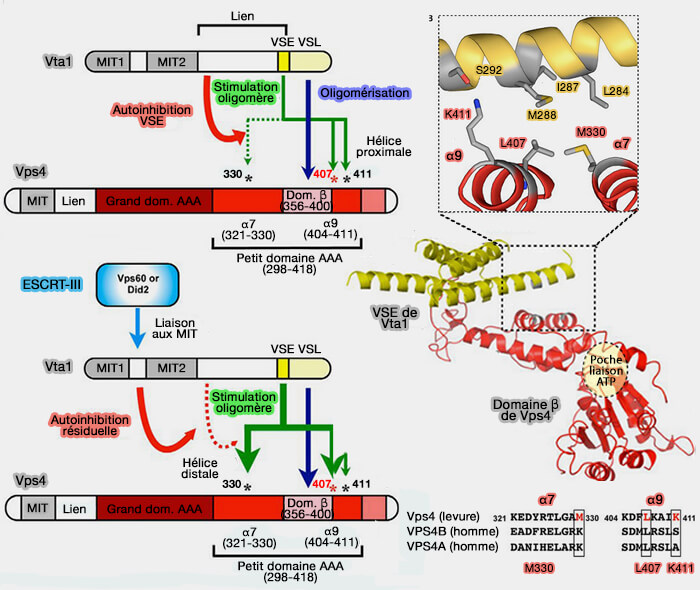
(Figure : vetopsy.fr d'après Davies et coll)
La structure du complexe du domaine β LIP5 VSL-VPS494 a conduit à un modèle dans lequel un réseau continu de dimères LIP5 connecte un réseau d'hexamères VPS4 (Interactions of the human LIP5 regulatory protein of the Vps4-Vta1 Interface in ESCRT-III Recycling 2010).
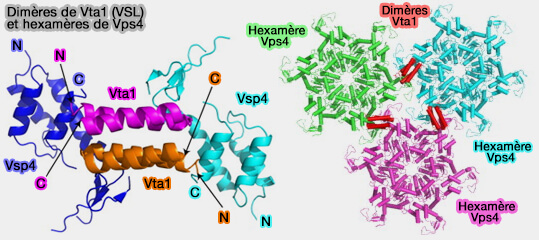
(Figure : vetopsy.fr d'après Yang et Huxley)
2. CHMP3 se lie aux enzymes de déubiquitylation (DUB) :
- AMSH, désignée aussi STAMBP (Structural Basis for ESCRT-III CHMP3 Recruitment of AMSH 2011).
- UBPY, dénommée aussi USP8.
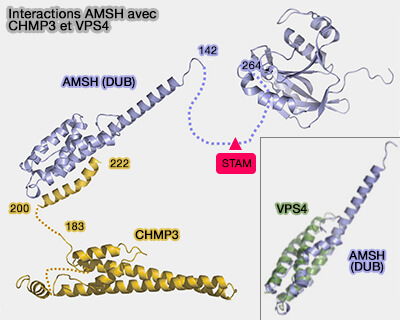
Interactions AMSH avec CHMP3 et VPS4
(Figure : vetopsy.fr d'après Solomons et coll)
Ces DUB sont aussi liées à la sous-unité STAM de ESCRT-0 et on ne sait pas si leurs recrutements sont indépendants ou non.
Conséquences
L'engagement du domaine MIT de VPS4 par des motifs MIM active puissamment l'hydrolyse de l'ATP par le domaine catalytique de VPS4 (Vps4 Stimulatory Element of the Cofactor Vta1 Contacts the ATPase Vps4 α7 and α9 to Stimulate ATP Hydrolysis 2014 et Binding of Substrates to the Central Pore of the Vps4 ATPase Is Autoinhibited by the Microtubule Interacting and Trafficking (MIT) Domain and Activated by MIT Interacting Motifs (MIMs) Vps4-Substrate Interactions 2015).
1. Dans le modèle de l'hexamérisation asymétrique décrit plus haut dont il existe plusieurs modèles, Vps4 procède par transition entre les configurations des sous-unités autour de l'anneau hexamérique, i.e. transition de la configuration des sous-unités F, A, B, C, D, E aux configurations de sous-unités A, B, C, D, E, F.
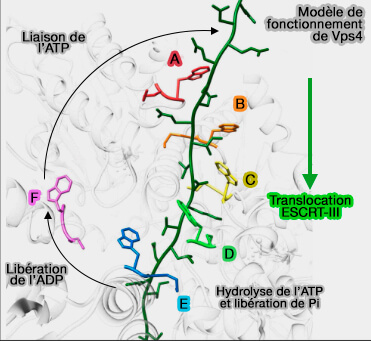
Modèle de fonctionnement de Vps4
(Figure : vetopsy.fr d'après Han
et Hill)
a. Vps4 " marche " le long des ESCRT-III par l'action des sous-unités qui :
- dénudent l'extrémité arrière de l'hélice formée par les cinq sous-unités, i.e. la sous-unité E,
- passent par la conformation transitoire, i.e. déplacement de la sous-unité F,
- permettent à l'extrémité de l'hélice, i.e. la sous-unité A, de se lier au dipeptide suivant dans la séquence ESCRT-III.
b. Conformément à ce modèle, le grand domaine ATPase de la sous-unité F est déplacé hors de l'axe de l'hélice, dégagé du peptide ESCRT-III.
- Il n'a que des contacts mineurs avec les grands domaines de sous-unités voisines Vps4.
- La sous-unité F est maintenue dans le complexe Vps4 principalement par des contacts plus périphériques des petits domaines d'ATPase.

L'avancement de l'hexamère Vps4 le long du polypeptide ESCRT-III étendu est équivalent au transport des ESCRT-III par les pores des hexamères Vps4, ce qui se traduira par le dépliage et le démontage d'ESCRT-III (Vps4 disassembles an ESCRT-III filament by global unfolding and processive translocation 2015).

Modèle de translocation de ESCRT-II
par Vps4 tiré de : Structure and mechanism of
the ESCRT pathway AAA+ ATPase Vps4
(Hann et all - 2017 -) à voir absolument !
2. Une caractéristique intéressante de ce modèle est que chaque sous-unité Vps4 n'a besoin d'adopter qu'une conformation et que son dipeptide ESCRT-III associé passe à travers le pore de la position de la sous-unité A à la position de la sous-unité E.
- Il semblerait que l'interface des sous-unités EF soit plus ouverte que les autres, ce qui favoriserait l'hydrolyse de l'ATP à l'interface DE, i.e. provoquant la déstabilisation des interactions et une conformation EF plus lâche.
- Vps4 n'aurait besoin de déplier qu'environ la moitié des sous-unités d'un filament afin d'en démonter l'intégralité.
3. Quand l'affinité des CHMP pour Vps4 est faible (CHMP4) ou négligeable (CHMP3), on n'a pas encore explicité comment se fait le recyclage.
- Les sous-unités à affinité forte (CHMP1, CHMP2, CHMP6 et IST1) aident les faibles à se dissocier.
- Lip5/Vta1 peut aider ce désassemblage.
Une fois le recyclage des sous-unités à affinité forte effectué, tout s'effondre.
Remodelage et fission
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeMitochondriesSystème endomembranaireRéticulum endoplasmiqueAppareil de GolgiEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaireMoteurs moléculairesVoies de signalisation


