Fusion membranaire : protéines SNARE
Vue d'ensemble et structure
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Transport membranaire
- Transports membranaires sans mouvements membranaires
- Transports membranaires avec mouvements membranaires
: trafic vésiculaire
- Fusion/fission membranaire
- Fusion membranaire
- Vue d'ensemble de la fusion membranaire
- Machinerie de fusion
- Mécanisme de fusion membranaire
- Fusion des différents compartiments membranaires
- Fission membranaire
- Fusion membranaire
- Endocytose
- Voie sécrétoire
- Fusion/fission membranaire
- Protéines membranaires
- Moteurs moléculaires
- Voies de signalisation

Les protéines SNARE catalysent les réactions de fusion membranaire au cours du trafic vésiculaire dans le système endomembranaire.
Généralités
1. Le trafic vésiculaire dans les cellules eucaryotes est effectué par toutes sortes de vésicules de transport qui bourgeonnent à partir d'un compartiment précurseur et sont transportées vers leur compartiment de destination où elles s'amarrent et fusionnent (
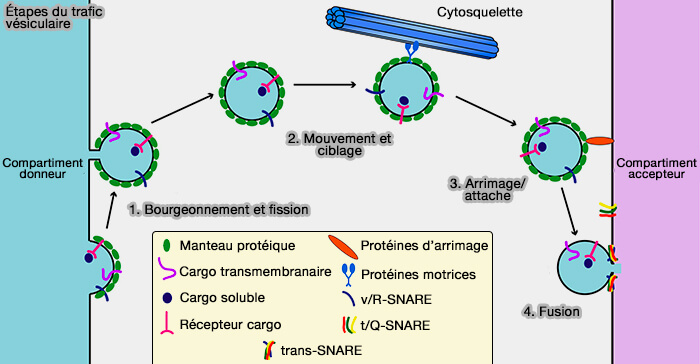
(Figure : vetopsy.fr d'après Cai et coll)

La fusion des différents organites est étudiée dans des chapitres spéciaux (
2. Lorsqu'une vésicule est libérée de son compartiment donneur, elle doit contenir un " plan de navigation " intégré afin d'atteindre la bonne destination intracellulaire, i.e. une sorte de code zip.
Pour atteindre leur destination intracellulaire, les vésicules de transport doivent comporter des signaux de ciblage comme les petites GTPases Rab et les phosphoinositides qui sont reconnus par des facteurs de liaison dans le cytoplasme et qui relient les vésicules à leur compartiment de destination respectif (
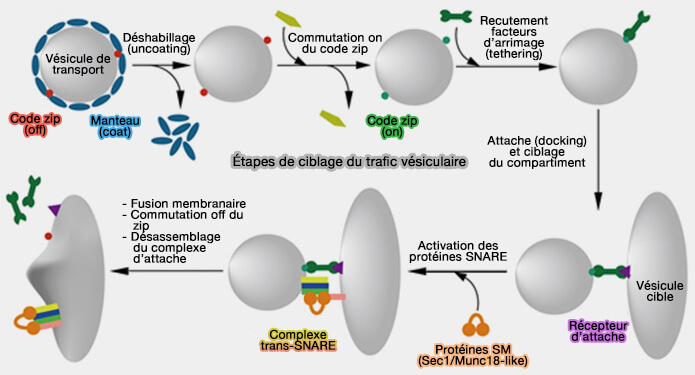
(Figure : vetopsy.fr d'après Koike et Reinhard)

La dernière étape de la fusion est effectuée par les protéines SNARE (The Multifaceted Role of SNARE Proteins in Membrane Fusion 2017).
Remarque : une exception partielle est celle de la fusion homotypique des membranes du réticulum endoplasmique (RE) par les atlastines des métazoaires/Sey1p des levures (Fusion of the endoplasmic reticulum by membrane-bound GTPases 2016).
Vue d'ensemble des protéines SNARE
La famille des protéines SNARE (Soluble N-ethylmaleimide-sensitive factor Attachment protein REceptor) est composée de 36 membres chez l'homme, 25 membres chez la levure qui résident sur toutes les endomembranes (An Elaborate Classification of SNARE Proteins Sheds Light on the Conservation of the Eukaryotic Endomembrane System 2007).

Vous pouvez lire : Structure and function of SNARE and SNARE-interacting proteins (2005).
1. Les protéines SNARE forment quatre sous-familles conservées au cours de l'évolution :
- les v-SNARE (v pour vésiculaire), qui sont pour la plupart des R-SNARE,
- les t-SNARE (t pour target, cible), qui sont pour la plupart des Q-SNARE, comprennent les Qa, Qb et Qc (
infos sur la nomenclature).
2. La catalyse de la fusion membranaire par les protéines SNARE est médiée par l'assemblage progressif en forme de fermeture éclair de quatre motifs SNARE, un membre en général de chaque sous-famille (Qa/b/c et R) résidant sur les deux membranes opposées et formant le complexe trans-SNARE (SNARE complex assembly and disassembly 2018).
- Les protéines SNARE ne forment qu'un nombre défini de complexes SNARE compétents en fusion.
- La fermeture éclair est exergonique et entraîne la formation d'un faisceau de quatre hélices avec les domaines transmembranaires adjacents les uns aux autres, tirant ainsi les membranes ensemble et surmontant la barrière d'énergie pour la fusion (SNARE zippering 2016).
3. Après la fusion, l'ATPase NSF (N-ethylmaleimide Sensitive Factor) et les α-SNAP (Soluble NSF Attachment Protein), i.e. qui n'ont rien à voir avec les Q-SNARE comme SNAP-25 par exemple, désassemblent le complexe SNARE avec l'énergie libérée lors de l'hydrolyse de l'ATP, rendant les SNARE individuels disponibles pour les prochains cycles de fusion (
Structure des protéines SNARE
Les SNARE sont des petites protéines, de structure et de taille variées, qui contiennent au moins un motif SNARE de 60 à 70 résidus (Tethering the assembly of SNARE complexes 2013).
Domaine ou motif SNARE
Vue d'ensemble
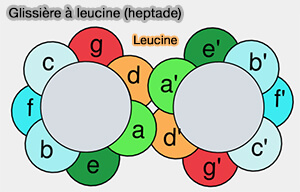
(Figure : vetopsy.fr)
1. Le domaine SNARE, i.e. moteur crucial de la fusion, est composé de huit répétitions de 7 acides aminés caractéristiques (A-G), qui peuvent former des structures coiled coil (Crystal structure of the endosomal SNARE complex reveals common structural principles of all SNAREs 2002).
- A et D sont des résidus hydrophobes, appelés aussi couches (layers), et forment des interactions étroites.
- Les autres résidus sont polarisés ou chargés.
Remarque : on retrouve souvent ces heptades dans des molécules comme la tropomyosine (

2. Le domaine SNARE peut être divisé en deux domaines, i.e. N-terminal (NTD) et C-terminal (CTD).
Remarque : SNAP-25, 23, 29, 47 possèdent deux motifs SNARE.
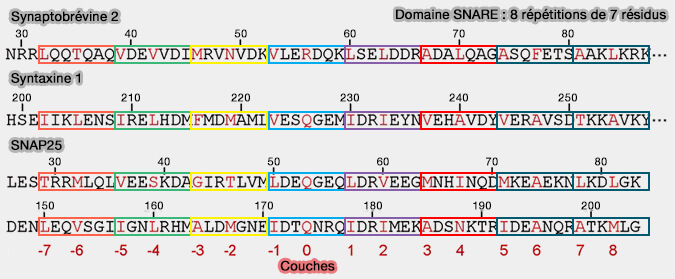
(Figure : vetopsy.fr)
Couche 0
1. Au milieu du domaine SNARE, il y a un seul résidu polaire ou chargé qui réside dans la position D, en général une glutamine (Q) ou une arginine (R), connue sous le nom de couche 0 (Crystal structure of a SNARE complex involved in synaptic exocytosis at 2.4 A resolution 1998).
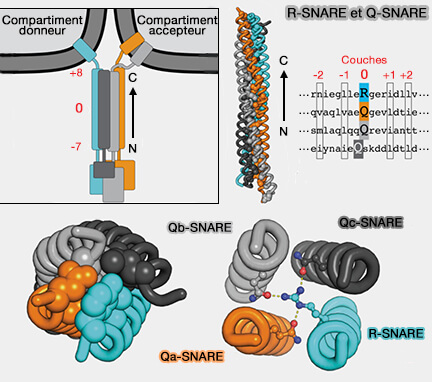
(Figure : vetopsy.fr d'après Baker et coll)
2. Ces résidus de couche zéro, qui sont presque universellement conservés parmi les complexes SNARE, ont donné lieu à une nomenclature alternative utile (Conserved structural features of the synaptic fusion complex: SNARE proteins reclassified as Q- and R-SNAREs 1998 et An Elaborate Classification of SNARE Proteins Sheds Light on the Conservation of the Eukaryotic Endomembrane System 2007).
a. Les R-SNARE, qui contiennent une arginine (R), sont pour la plupart des v-SNARE (v pour vésiculaire) et comprennent plusieurs familles.
b. Les Q-SNARE, qui contiennent une glutamine (Q), sont pour la plupart des t-SNARE (t pour target, cible) qui comprennent les Qa, Qb et Qc, i.e. a, b et c indiquant leurs positions relatives dans le faisceau à quatre hélices.

Les R-SNARE et les Q-SNARE, ainsi que leurs différents membres, sont étudiées dans des chapitres spéciaux.
Autres domaines
Lien et domaine transmembranaire
1. Dans la plupart des SNARE, le motif SNARE est relié par un lien (linker ou LD), i.e. 147-187 dans la syntaxine-1 (Stx1) qui comprend un motif juxtamembranaire (JM) à un domaine d'ancrage C-membranaire terminal (TM ou TMD).
Le ciblage, souvent unique sur une membrane, i.e. organites du système endomembranaire, mitochondries, membrane plasmique, peroxysomes, est accompli en modifiant :
- soit la composition des résidus d'acides aminés C-terminaux qui touchent le domaine SNARE,
- soit la longueur du domaine transmembranaire.
Les TMD ont des fonctions mécaniques qui vont au-delà de l'ancrage passif du complexe SNARE générateur de force à la fusion des membranes.

Le rôle des JM et des TMD des v-SNARE est essentiel à la fusion membranaire (
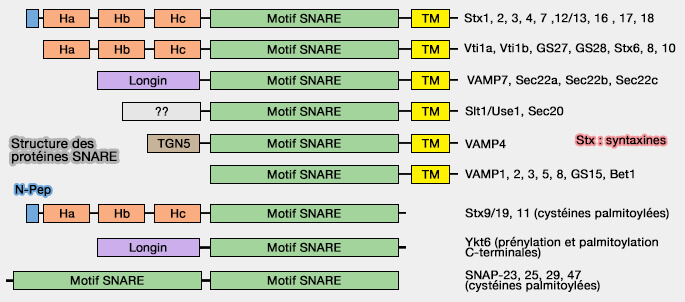
(Figure : vetopsy.fr d'après Hong et Lev)
2. Toutefois, SNAP-25 et 6 autres SNARE (
Le remplacement du domaine transmembranaire par des ancres lipidiques conduit à une étape intermédiaire de fusion membranaire où seuls les deux feuillets en contact fusionnent et non les deux feuillets distaux des deux bicouches membranaires (Mechanisms of membrane fusion: disparate players and common principles 2008).
Par exemple, dans le complexe SNARE neuronal (
- les motifs SNARE de la syntaxine 1A (Stx1A) et de la synaptobrévine 2/VAMP2 sont reliés par de courts liens (linkers) aux régions transmembranaires C-terminales.
- SNAP-25 est ancré à la membrane plasmique via des chaînes palmitoyle liées à des résidus de cystéine dans une région de boucle reliant ses deux motifs SNARE, appelées sn1 et sn2.
Régions N-terminales régulatrices
(NRD ou N-terminal Regulatory Domain)
De nombreuses SNARE possèdent des régions N-terminales régulatrices (NRD ou N-terminal Regulatory Domain).
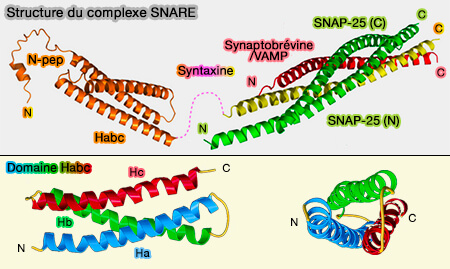
(Figure : vetopsy.fr d'après Stepien et coll)
1. Le peptide N-terminal de Stx1 (résidus 1-26), 5 et 16 interagit avec les protéines SM - Sec1/Munc18-like - (SNAREs and traffic 2005).
2. Le domaine Habc de Stx1 (résidus 27-146) forme un faisceau de trois hélices α, relié au domaine SNARE (147-187) avec lequel il interagit pour inhiber la formation du complexe trans-SNARE (The Habc domain of syntaxin 3 is a ubiquitin binding domain 2020).
3. Le domaine longin de VAMP7 interagit avec le domaine SNARE (The binding of Varp to VAMP7 traps VAMP7 in a closed, fusogenically inactive conformation 2012 et Structure and function of longin SNAREs 2015).
4. L'extension N-terminale de VAMP4 (TGN5) contient un motif LL et des groupes acides qui contribuent au recyclage des endosomes vers le réseau trans-Golgi (TGN) (VAMP4 cycles from the cell surface to the trans-Golgi network via sorting and recycling endosomes 2007).
Formation du complexe SNARE
Biologie cellulaire et moléculaireConstituants de la celluleTransport membranaireTransports sans mouvements membranairesTransports passifsTransports actifsTransports avec mouvements membranaires : trafic vésiculaireFusion/fission membranaireEndocytoseVoie sécrétoireCanaux ioniquesTransporteursUniportsPompesCo-transporteursMoteurs moléculairesVoies de signalisation


