Constituants cellulaires : protéasome 26S
Fonctionnement : désubiquitination et protéolyse du substrat
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Pendant la translocation du substrat dans la chambre catalytique, plusieurs processus arrivent concomitamment :
- le dépliage du substrat à travers l'anneau OB ou N,
- le clivage de l'ubiquitine qui peut être alors recyclée,
- la protéolyse du substrat en peptides courts de 7 à 8 acides aminés.
Désubiquitination du substrat
La désubiquitination du substrat se produit en même temps que la translocation et dépend du cycle ATPasique.
Le protéasome ne dégrade pas les ubiquitines (au moins 4) du substrat, mais il les clive pour les recycler.
Rpn11
Rpn11, une désubiquitinase (DUB) de la famille des MNP/JAMM, est au coeur de ce processus.
(Figure : vetopsy.fr d'après Lander)
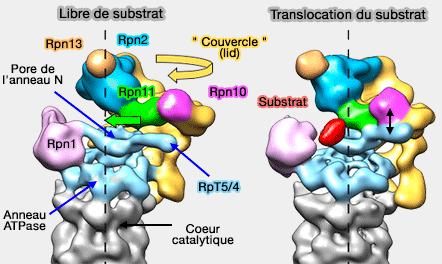
1. Dans les protéasomes libres de tout substrat, Rpn11 est situé sur le côté de l'anneau OB de la base, au-dessus de la superhélice des Rpt5/4.
- Ce positionnement prévient l'activité de son site catalytique.
- Par contre, le pore de l'anneau OB ou N est accessible à l'extrémité souple du substrat attaché à un récepteur d'ubiquitine.
2. Lors du déplacement du " couvercle " dans l'engagement du substrat, Rpn11 se décale de 18 Å, si bien que son site catalytique se positionne face au canal central (Conformational switching of the 26S proteasome enables substrate degradation 2013)
Ce processus est ATP-dépendant et est couplé à la translocation du substrat dans la chambre catalytique, qui automatiquement positionne correctement la chaîne d'ubiquitine et le site catalytique : ce processus prévient le retrait prématuré de l'ubiquitine à partir d'un substrat qui n'est pas encore engagé.
DUB supplémentaires
Des DUB supplémentaires peuvent s'associer de manière réversible au protéasome.
- Elles semblent attaquer les chaînes d'ubiquitine indépendamment de la dégradation du substrat, les raccourcissant plutôt que les enlevant entièrement.
- Ceci pourrait contribuer à la discrimination par le protéasome de courtes ou de longues chaînes de polyubiquitines, limiter l'action du protéasome sur ses substrats ou réguler leur dégradation.
(Figure : vetopsy.fr d'après Bashore et Vanderlinden)
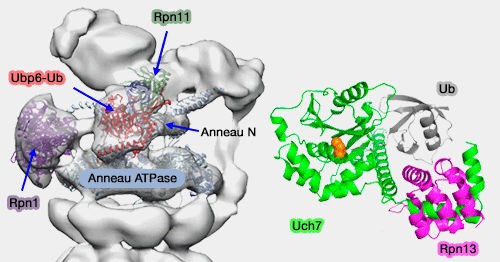
1. Ubp6 de la levure interagit avec la base du protéasome.
- Elle contacte, par son domaine UBL, Rpn1, l'anneau OB et l'anneau ATPase (en particulier le domaine N-terminal de Rpt1), directement en face de Rpn11.
- Ubp6, lié à l'ubiquitine, semble inhiber la désubiquitination du substrat par Rpn11, stabilise la conformation du substrat avec le protéasome et interfère avec l'engagement ultérieur d'un substrat.
-
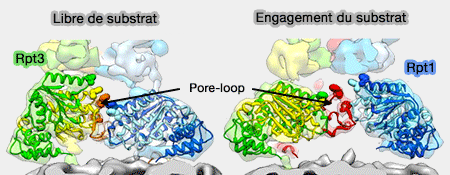
Repositionnement des Rpt lors de l'engagement du substrat
(Figure : vetopsy.fr d'après Lander)
2. Uch37 des mammifères se retrouve aussi dans INO80, régulateur de la transcription et la réparation d'ADN (Distinct Modes of Regulation of the Uch37 Deubiquitinating Enzyme in the Proteasome and in the Ino80 Chromatin-Remodeling Complex 2008).
Le domaine C-terminal de Rpn13 se lie au domaine C-terminal autoinhibiteur de Uch37, qui est lié par son domaine N-terminal à Rpn2 (Structural Basis for the Activation and Inhibition of the UCH37 Deubiquitylase 2014).
3. USP14 des mammifères aurait un rôle similaire (Enhancement of Proteasome Activity by a Small-Molecule Inhibitor of Usp14 2010).
4. Hul5/KIAA10, une E3 ubiquitine ligase HECT, qui déploie les chaînes d'ubiquitine sur le protéasome, pourrait coopérer avec ces DUB, mais semble aussi jouer un rôle inhibiteur sur le protéasome en polyubiquitinant Rpn13 (Ubiquitin Chains Are Remodeled at the Proteasome by Opposing Ubiquitin Ligase and Deubiquitinating Activitie 2006 et The Ubiquitin Ligase Hul5 Promotes Proteasomal Processivity 2009) et Autoubiquitination of the 26S Proteasome on Rpn13 Regulates Breakdown of Ubiquitin Conjugates 2014).
Protéolyse du substrat
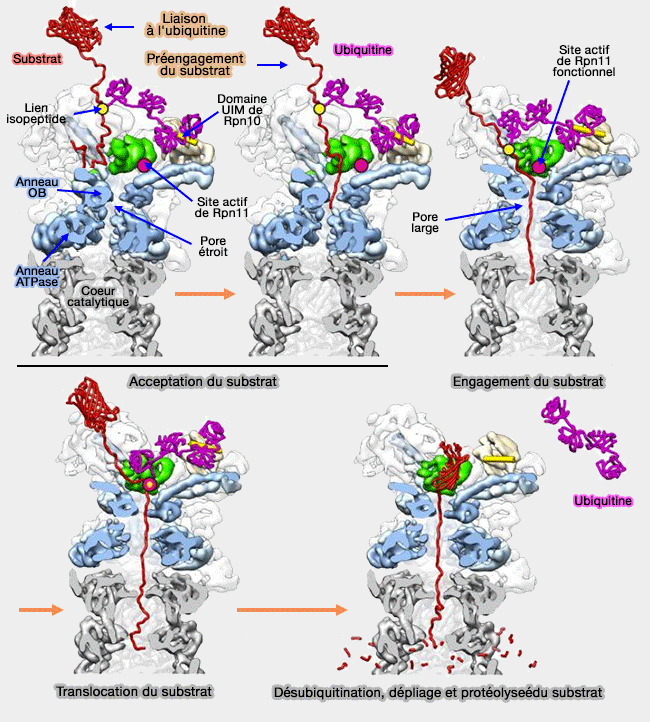
(Figure : vetopsy.fr d'après Lander)
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


