Constituants cellulaires : protéasome 26S
Fonctionnement : engagement du substrat
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Une fois l'acceptation du substrat effectuée, le protéasome engage la chaîne polypeptidique du substrat, hydrolysant l'ATP en ADP (cycle ATPasique) dans les Rpt de la base de la particule 19S.
Ce cycle ATPasique permet :
- le déplacement des anneaux de la base, l'anneau OB (ou N) formé par les domaines OB et l'anneau ATPase formé par les domaines AAA+,
- l'alignement avec le pore de l'anneau α du coeur catalytique,
- l'élargissement du pore de l'anneau ATP qui s'élargit pour passer de 9 Å de diamètre à 12 à 20 Å pour l'engagement du substrat.

Le fonctionnement détaillé du protéasome qui suit est tiré en grande partie de l'article Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome 2014, mais aussi de nombreux autres.…
Vous pouvez aussi voir deux films tirés de cet article : sur les changements de conformation vus de côté et vus du haut du protéasome.
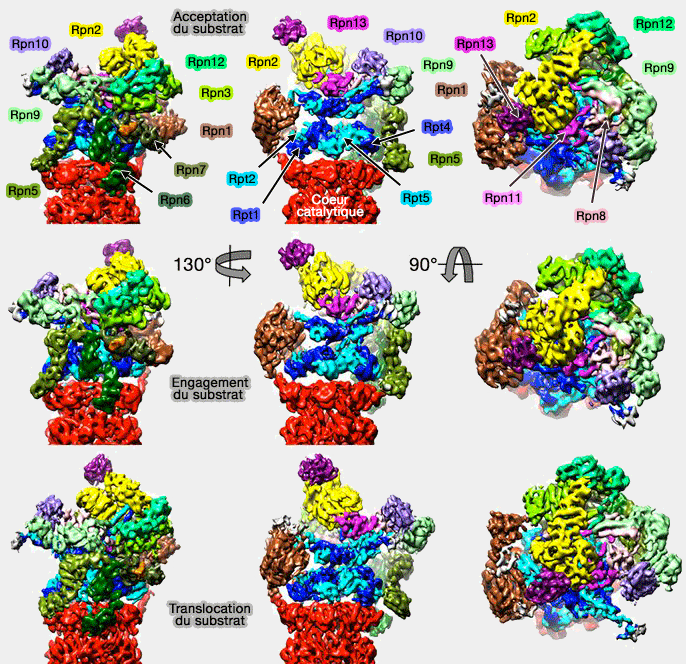
(Figure : vetopsy.fr d'après Unverdorben)
Engagement du substrat
Déplacement du " couvercle " (lid)
Le " couvercle " (lid), avec Rpn2 et Rpn10, se déplace drastiquement par rapport au module AAA en subissant une rotation de 25°, ce qui provoque des changements réarrangements majeurs.
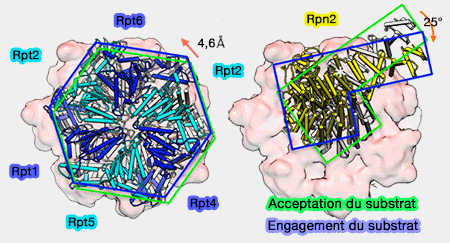
(Figure : vetopsy.fr d'après Unverdorben)
Cette rotation serait liée à celle de Rpn2.
- Rpn10 est lié à Rpn11, lui-même lié à Rpn2. Rpn2 est lié à Rpn12 et à Rpn3 et au faisceau des extrémités C-terminales des sous-unités du " couvercle ".
- Le mouvement de Rpn2 se traduit par une rotation de 25° degrés du couvercle autour du point d'ancrage de Rpn2 au domaines coiled-coil (superhélice) des Rpt3/6 (cf. alignement du substrat).
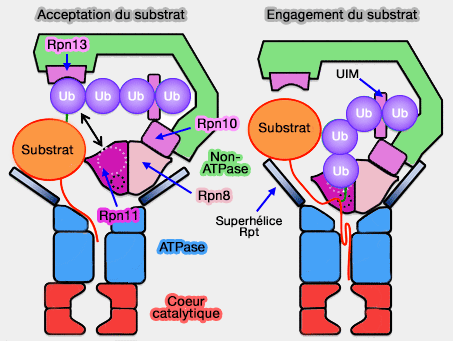
1. Le repositionnement concomitant de Rpn10 le place alors idéalement au-dessus de Rpn11, la désubiquitinase (DUB) de la famille MPN/JAMM, formant un dimère avec Rpn8 du " couvercle " (lid), pour lui transférer le substrat polyubiquitiné.
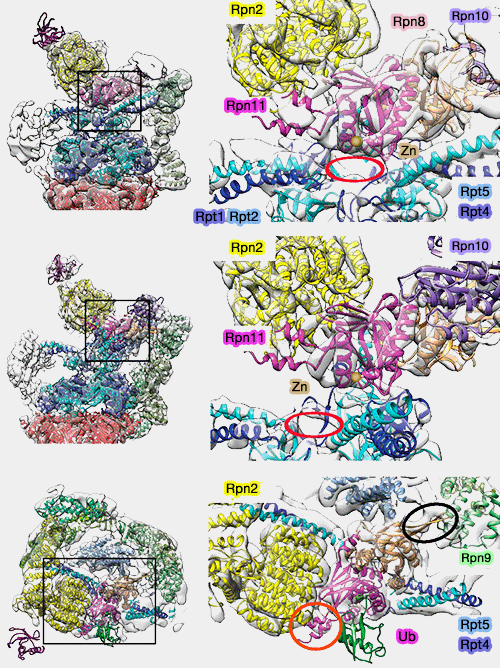
(Figure : vetopsy.fr d'après Pathare)
La liaison simultanée de la chaîne de polyubiquitine à Rpn10 et Rpn11 pourrait ainsi expliquer la préférence du protéasome pour les substrats polyubiquitinés.
2. Cette rotation provoque une translation de 25Å du dimère Rpn11/8 qui positionne le site actif de Rpn11 dans l'axe de l'anneau OB (ou N).
- L'anneau OB et l'insertion 2 de Rpn11 forment une cage étroite autour de Rpn11.
- Le site de liaison avec l'ubiquitine est accessible : l'espace pour accueillir le substrat est élargi, libérant alors le site de désubiquitination.
- Les contacts des autres sous-unités du " couvercle " ne changent pas : en particulier, Rpn10 est toujours positionné par rapport à la superhélice (coiled coil) des Rpt4/5 et de l'anneau N.
Les mouvements complexes de Rpn11/8 sont expliqués dans Crystal structure of the proteasomal deubiquitylation module Rpn8-Rpn11 2014 (voir en particulier les figures supplémentaires).
Déplacement de la base
L'anneau ATPase est positionné différemment : translaté (≅5Å), en rotation latérale (≅1°), et moins incliné (≅2°) par rapport à l'anneau OB (ou N).
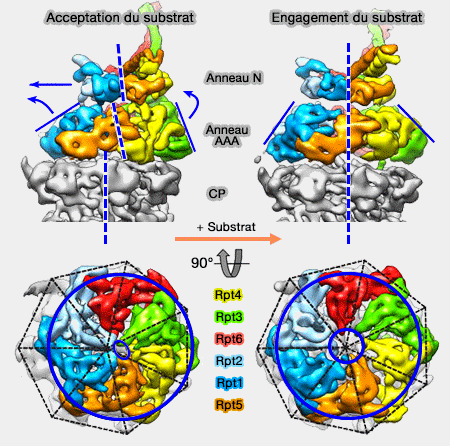
(Figure : vetopsy.fr d'après Bhattacharyya)
1. Cette transformation positionne les Rpt (2, 6 et 3) plus loin du coeur catalytique, et Rpt2 est proche de l'extrémité N-terminale de Rpn1.
D'une part, les interfaces entre les domaines AAA deviennent très uniformes autour de l'anneau.
- Cette conformation est proche de ceux des corps rigides (Rigid Bodies) de ClpX, formé par le grand sous-domaine et le petit sous-domaine de l'unité AAA adjacente .
- Dans un protéasome libre de tout substrat, il semble qu'un corps rigide soit déjà formé entre le petit sous-domaine de Rpt3 et le grand sous-domaine de Rpt4. Comme Rpt3 et Rpt4 sont situés au sommet de la spirale formée par les Rpt, elles pourraient être les premières à interagir avec ce dernier, et entraîner les autres dans le mouvement.
- La liaison du nucléotide modifie la rotation entre les grands et les petits domaines d'une sous-unité, et expliquent le mécanisme permettant de relier le cycle ATPasique aux changements conformationnels dans l'anneau hexamérique des Rpt, à l'alignement des anneaux et, enfin à la translocation du substrat.
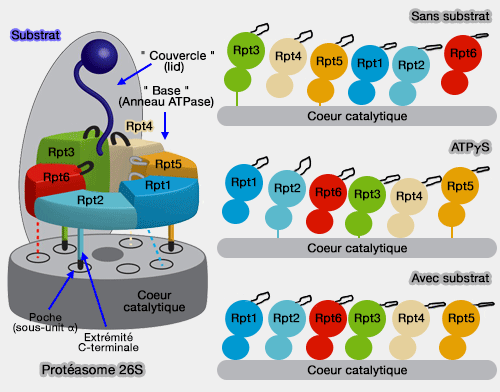
(Figure : vetopsy.fr d'après Estin et Bhattacharyya)
D'autre part, la spirale est modifiée.
- Dans un protéasome libre de tout substrat, la spirale formée par les grands sous-domaines des Rpt les placent à des distances différentes du pore central, Rpt3 étant le plus éloigné, Rpt2 le plus proche. Rpt6 sert de liaison entre ces deux sous-unités pour fermer la spirale : il sert aussi de point d'ancrage pour assurer la bonne position de l'anneau ATPase au sommet de l'anneau α.
- Par contre, lorsque le substrat s'engage, le basculement provoque un nouvel arrangement des Rpt : Rpt1 est positionné le plus haut et Rpt4 le plus près et c'est alors Rpt5 qui ferme la spirale.
2. Rpn1 lui-même est translaté et tourne d'environ 35° pour positionner son domaine N/C, formé par ses extrémités N- et C-terminales, plus près de l'anneau ATPase et du coeur catalytique.
(Figure : vetopsy.fr d'après Pathare)
3. Les contacts dans les domaines N-terminaux de Rpn5 et Rpn6 se modifient par le mouvement de la base et de Rpt3 qui les éloignent de leur domaine PCI - 30Å et 25 Å respectivement - (Conformational switching of the 26S proteasome enables substrate degradation 2013).
- Rpn5 ne contacte plus l'anneau d'ATPase alors que son interface avec α1 s'agrandit.
- Rpn6 conserve ses contacts avec les anneaux ATPases et α, mais les interactions sont différentes.
Les contacts entre Rpn7 et Rpt6 restent constants.
4. La courbure de la superhélice des Rpt6/3 change aussi.
- Les contacts avec le " couvercle " et Rpn2 sont plus étendus.
- La superhélice de Rpt4/5 contacte Rpn10.
Translocation du substrat
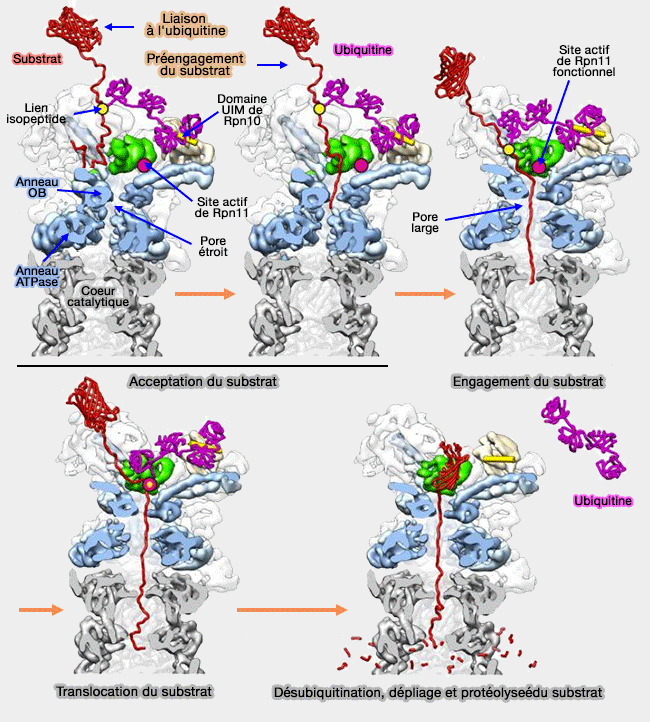
(Figure : vetopsy.fr d'après Lander)
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


