Constituants cellulaires : protéasome 26S
Fonctionnement : acceptation du substrat
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

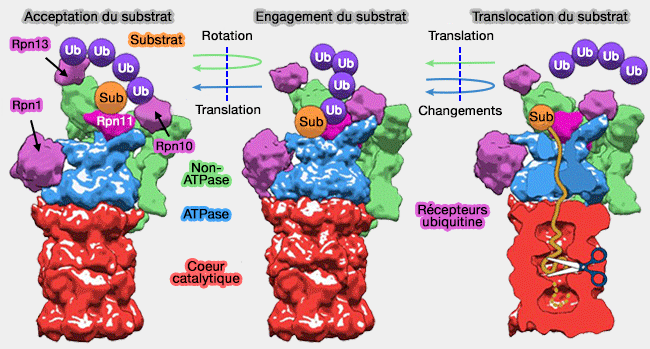
Le protéasome 26S reconnaît, en général, des protéines polyubiquitinées dont la chaîne comprend au moins 4 ubiquitines et plus par la particule 19S
La première est liée à la lysine du substrat, la deuxième à une lysine particulière (polyubiquitination)…
(Figure : vetopsy.fr d'après Unverdorben)
La première étape du fonctionnement du protéasome est la liaison de la chaîne d'ubiquitine à ses récepteurs (Degradation signal diversity in the ubiquitin-proteasome system 2009 et The Ubiquitin–Proteasome System of Saccharomyces cerevisiae 2012)..
Dans un protéasome qui accepte le substrat,
- les anneaux de la base, l'anneau OB (ou N) formé par les domaines OB et l'anneau ATPase formé par les domaines AAA+, et l'anneau α du coeur catalytique ne sont pas alignés (10 Å).
-
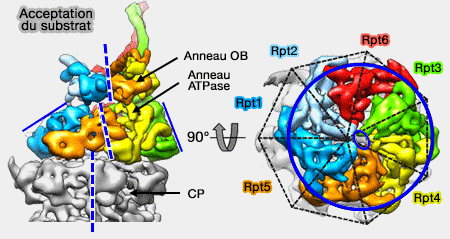
Positionnement des anneaux
(Figure : vetopsy.fr d'après Bhattacharyya)
Acceptation
du substrat
Le protéasome reconnaît les chaînes d'ubiquitine grâce à (Recognition and Processing of Ubiquitin-Protein Conjugates by the Proteasome 2009) :
- Rpn10 et Rpn13, principalement,
- d'autres récepteurs alternatifs d'ubiquitine et détachables dans certains cas.
Rpn10 et Rpn13
Rpn10, par son domaine UIM, et Rpn13, par son domaine PRU, sont des récepteurs de l'ubiquitine.
-
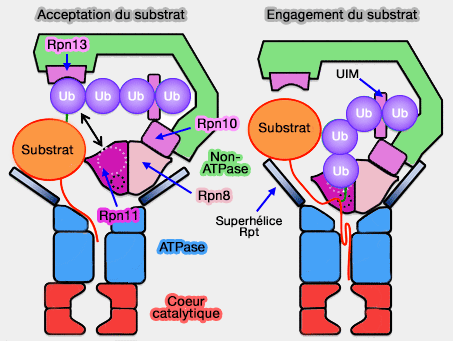
Modifications lors de l'acceptation et l'engagement du substrat
(Figure : vetopsy.fr d'après Pathare) - La liaison simultanée aux sous-unités Rpn10 et Rpn13 augmenterait l'affinité pour les chaînes d'ubiquitine.
- En outre, cela permettrait de ne pas se lier à une protéine monoubiquitinée utilisée le plus souvent comme signal.
Rpn10, situé à l'interface entre la base et le " couvercle ", est placé à proximité de Rpn11, la désubiquitinase (DUB) de la famille MPN/JAMM du " couvercle " (lid).
- Le site actif de Rpn11, est positionné au-dessus de la partie inférieure de la superhélice (coiled coil) de Rpt4/5, à 20 Å de l'axe de l'anneau OB.
- Le site de liaison à l'ubiquitine est accessible à l'extrémité souple du substrat, mais est trop étroit pour qu'une ubiquitine liée au substrat puisse atteindre le site actif de Rpn11.
- Les mouvements complexes de Rpn11/8 sont expliqués dans Crystal structure of the proteasomal deubiquitylation module Rpn8-Rpn11 2014 (voir en particulier les figures supplémentaires).
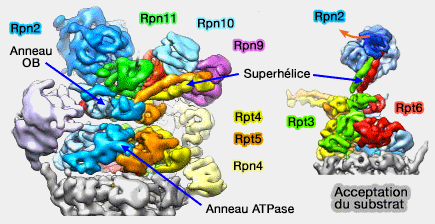
(Figure : vetopsy.fr d'après Bhattacharyya)
Rpn2 est en contact avec la superhélice (coiled coil) de Rpt3/6.
Les domaines PCI des Rpn du " couvercle " (lid) contactent les anneaux.
- Rpn5 et Rpn6, par leur répétition tétratricopeptide (TPR : tetratricopeptide repeat) N-terminale interagissent, respectivement, avec Rpt6 et Rpt3 de l'anneau ATPase et les sous-unités α1 et α2 du coeur catalytique.
- Rpn7 interagit uniquement avec Rpt6.
Récepteurs alternatifs d'ubiquitine
Des récepteurs alternatifs à l'ubiquitine contiennent des domaines UBL (UBiquitine-Like domain) et UBA (UBiquitin Associated).
-
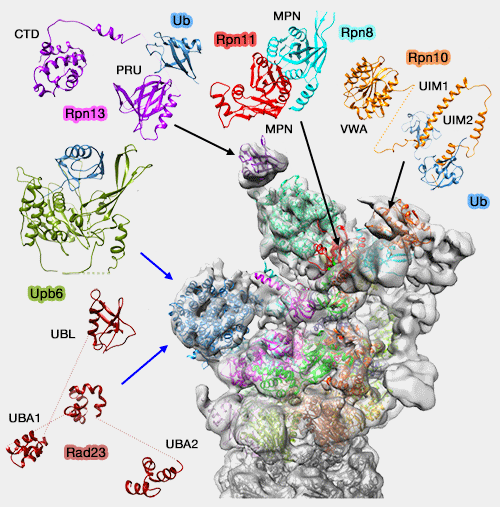
Protéines associées au protéasome pour la désubiquitination
(Figure : vetopsy.fr d'après Kish-Trier) - DSK2 (Proteasome subunit Rpn1 binds ubiquitin-like protein domains 2002),
- Ddi1 (Identification of a functional docking site in the Rpn1 LRR domain for the UBA-UBL domain protein Ddi1 2014).
Ces récepteurs se lient à Rpn1, la grande sous-unité organisatrice de la base du protéasome par leur domaine UBL et au substrat par leur domaine UBA.
- Leur liaison est complexe et demande des études complémentaires (Identification of a functional docking site in the Rpn1 LRR domain for the UBA-UBL domain protein Ddi1 2014).
- Leurs domaines sont connectés par un lien flexible qui permet la bonne orientation du substrat. Ainsi, les régions non structurées de l'extrémité du substrat atteignent les pales du moteur de translocation qui les feront entrer dans la chambre catalytique pour leur dégradation (Substrate selection by the proteasome during degradation of protein complexes 2009 et Together, Rpn10 and Dsk2 can serve as a polyubiquitin chain-length sensor 2009).
Engagement du substrat
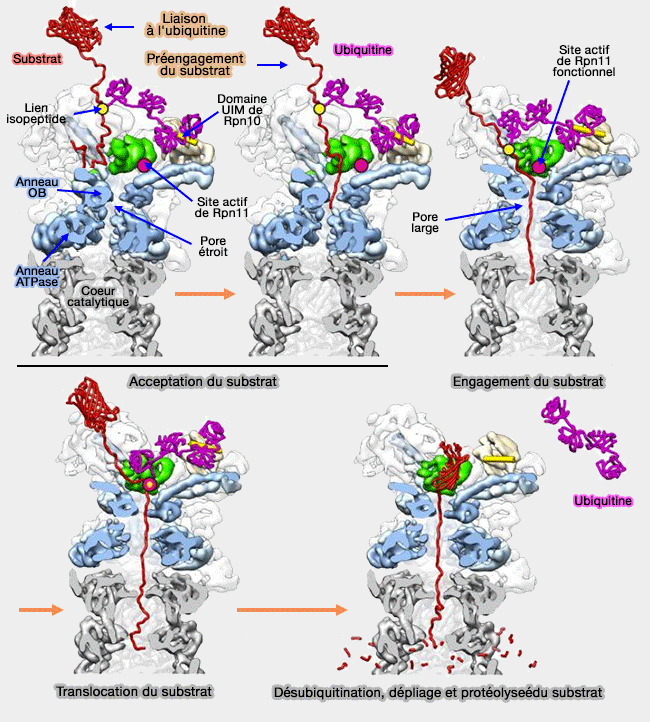
(Figure : vetopsy.fr d'après Lander)
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


