Constituants cellulaires : protéasome 26S
Fonctionnement : vue d'ensemble
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
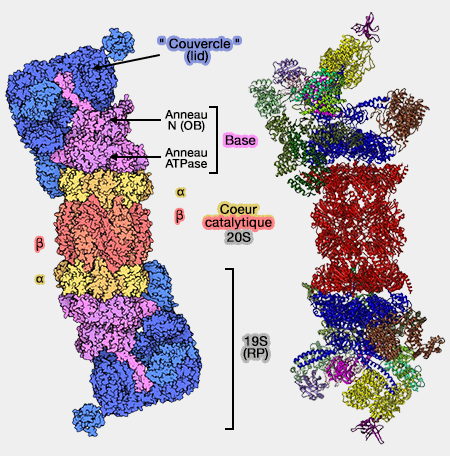
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les protéasomes sont des complexes enzymatiques multiprotéiques dont le rôle est de découper les protéines (protéolyse) en peptides.
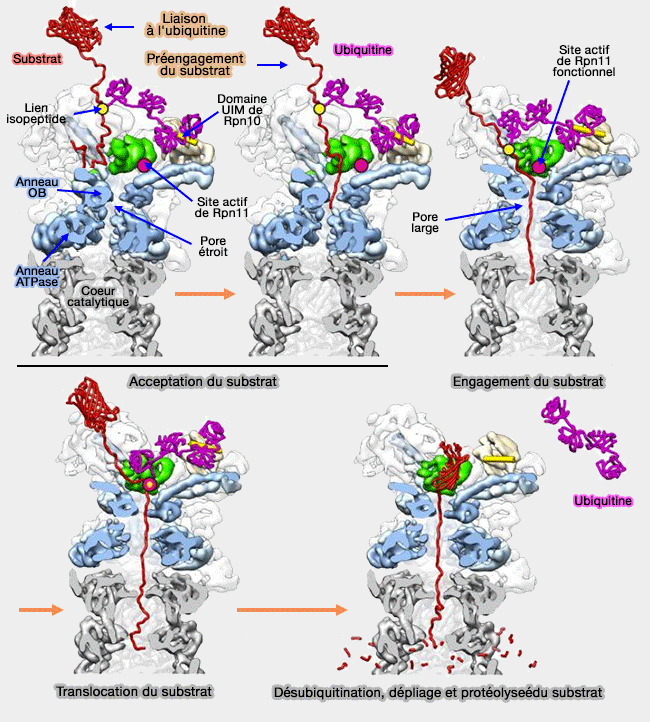
Le fonctionnement du protéasome se fait en plusieurs étapes donc certaines sont simultanées.
1. Le protéasome reconnaît, en général, des protéines polyubiquitinées dont la chaîne comprend au moins 4 ubiquitines et plus par la particule 19S (cf. acceptation du substrat)
La première est liée à la lysine du substrat, la deuxième à une lysine particulière (polyubiquitination)…
(Figure : vetopsy.fr d'après Lander)
Une fois l'acceptation du substrat effectuée, le processus est lancé. Les événements ne se suivent pas chronologiquement, mais s'effectuent
2. Le protéasome déplace la chaîne polypeptidique du substrat (cf. engagement du substrat), hydrolysant l'ATP en ADP (cycle ATPasique) dans les Rpt de la base de la particule 19S.
3. Il transloque les protéines vers la chambre catalytique (cf. translocation du substrat).
4. Plusieurs processus arrivent alors concomitamment :
- Il déplie le substrat et clive l'ubiquitine qui peut être alors recyclée (cf. désubiquitination du substrat),
- Il coupe la protéine séquentiellement en peptides courts de 7 à 8 acides aminés (cf. protéolyse du substrat).
(Figure : vetopsy.fr)
Ce processus lui permet :
- de remodeler des complexes protéiques en dégradant que la sous-unité sur laquelle il initie la première dégradation, par exemple, en clivant la partie active des protéines-cibles ou un domaine activateur ou inhibiteur dans les voies de signalisation par exemple ;
- de dégrader les protéines.
Le fonctionnement a été d'abord élucidé pour le complexe régulateur 11S qui a l'avantage d'être constitué par un complexe héxamérique d'AAA+ identiques et qui, en outre, ne reconnaît pas l'ubiquitine.
Celui de la RP 19S est, en gros, similaire (The 1.9 Å Structure of a Proteasome-11S Activator Complex and Implications for Proteasome-PAN/PA700 Interactions 2005 et Mechanism of gate opening in the 20S proteasome by the proteasomal ATPases 2008 et Proteasomal AAA-ATPases: Structure and function 2012).
Le fonctionnement détaillé du protéasome est tiré en grande partie de l'article Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome 2014, mais aussi de nombreux autres.
Vous pouvez aussi voir deux films tirés de cet article : sur les changements de conformation vus de côté et vus du haut du protéasome.
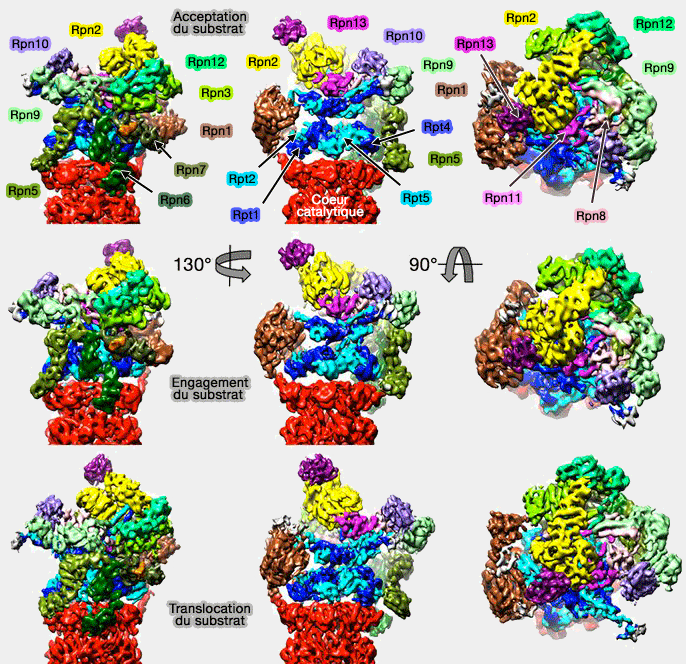
(Figure : vetopsy.fr d'après Unverdorben)
Acceptation du substrat
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


