Constituants cellulaires : protéasomes
Autres protéasomes
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

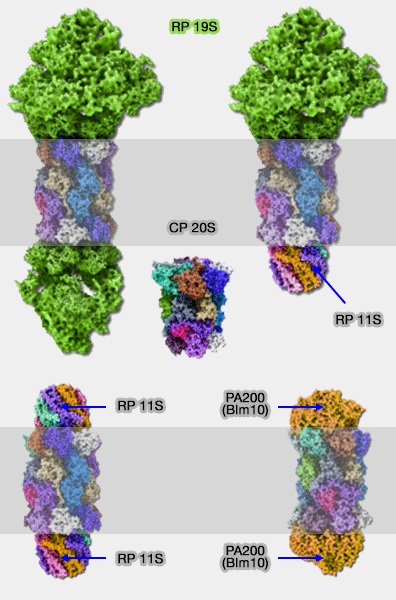
Les protéasomes, à part leur coeur catalytique 20S (CP : Core Particle), comporte en général un complexe régulateur 19S (RP : Regulatory Particle).
Toutefois, certains protéasomes remplacent ce complexe 19S (PA700) par un autre complexe régulateur (The proteasome: Overview of structure and functions 2009) :
- soit des deux côtés (PA200 ou 11S),
- soit d'un seul côté (11S) , sans que l'on sache bien comment il fonctionne.
PA est une abbréviation de Proteasome Activator.
PA200 /Blm10
(Figure : vetopsy.fr d'après Jung)
PA200 (Blm10 chez la levure, homologue à 20%), est une protéine monomérique nucléaire, impliquée dans les dommages à l'ADN ou certaines dégradations de protéines (Proteasomes Associated with the Blm10 Activator Protein Antagonize Mitochondrial Fission through Degradation of the Fission Protein Dnm1 2014).
Elle est présente dans les spermatoprotéasomes.
Complexes 11S
Les complexes 11S (REG ou PA28) peuvent être constitués de trois éléments α, β et γ, homologues à 50% environ.
Les complexes 11S sont composés de :
- 6 sous-unités identiques chez la levure, structure simple qui a permis de comprendre le fonctionnement des RP ;
- 6 ou 7 sous-unités (PA28αβ), uniquement dans le cytosol, sous forme α3β3, α4β3, α3β4,
- 7 sous-unités (PA28γ7) retrouvé dans le cytosol et le noyau. Ces derniers sont retrouvés dans les immunoprotéasomes.
Ces deux complexes (PA200 et 28) dégradent les protéines sans reconnaître l'ubiquitine contrairement au complexe 19S (Recognition and Processing of Ubiquitin-Protein Conjugates by the Proteasome 2009).
La protéine Cdc48/p97/VPC, qui est une protéine AAA+, peut aussi être utilisée comme complexe régulateur (Bipartite determinants mediate an evolutionarily conserved interaction between Cdc48 and the 20S peptidase 2013 et Cdc48: A Swiss Army Knife of Cell Biology 2013)
Assemblage du protéasome
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


