Constituants cellulaires : protéasome 26S
Assemblage du complexe régulateur 19S
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Le complexe régulateur 19S (RP : Regulatory Particle) est formé de deux parties, la base et le " couvercle ", qui s'assemblent indépendamment avant de s'associer.
Ce mode d'assemblage a l'avantage de limiter des assemblages aberrants entre " couvercle " et base et, en outre, d'interdire des activités ATPasiques ou de désubiquitination avant que le protéasome soit entièrement achevé.
Si l'assemblage du coeur catalytique est assez bien connu, celui du complexe régulateur l'est beaucoup moins.
Assemblage de la base
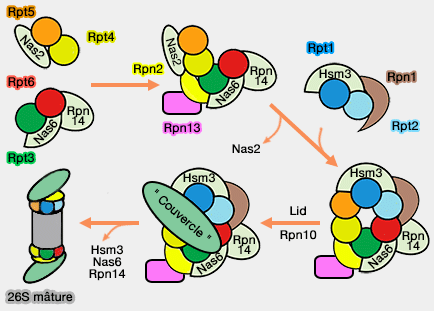
(Figure : vetopsy.fr d'après Tomko)
L'assemblage de la base est difficile à appréhender car les Rpt sont très proches dans leur structure : 120 arrangements différents pourraient être envisagés (Order of the proteasomal ATPases and eukaryotic proteasome assembly 2011).
RAC (RP Assembly Chaperones)
L'assemblage du complexe héxamérique des 6 Rpt (et Rpn1) nécessite 4 protéines chaperons différentes chez les levures (ou les mammifères).
Cet assemblage s'effectue par trois modules différents (Order of the proteasomal ATPases and eukaryotic proteasome assembly 2011) :
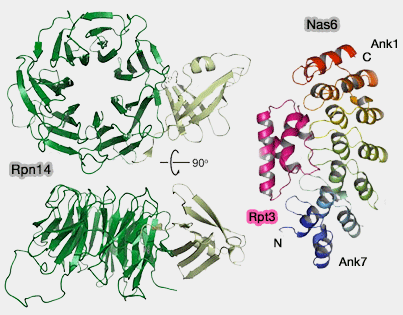
- Nas6 (p28/gankyrine) avec Rpt3 et Rpn14 (PAAF1 chez l'Homme) avec Rpt6 (Structure of the Oncoprotein Gankyrin in Complex with S6 ATPase of the 26S Proteasome 2007),
- Nas2 (p27) avecRpt4 et Rpt5,
- Hsm3 (S5b) avec Rpt1, Rpt2 et Rpn1 (Hsm3/S5b participates in the assembly pathway of the 19S regulatory particle of the proteasome 2009).
(Figure : vetopsy.fr d'après Takagi et Barrault)
La structure de ces RAC comprend des domaines d'interactions protéine/protéine qui reconnaîssent l'extrémité C-terminale du Rpt :
- Nas6, Rpt3, par ses 7 répétitions ankyrine (Structural basis for the recognition between the regulatory particles Nas6 and Rpt3 of the yeast 26S proteasome 2007) ;
- Rpn14, Rpt6, par ses 7 répétitions WD40 (Crystal Structure of Yeast Rpn14, a Chaperone of the 19 S Regulatory Particle of the Proteasome 2010),
- Nas2, Rpt5, par son domaine PDZ (Loss of Rpt5 Protein Interactions with the Core Particle and Nas2 Protein Causes the Formation of Faulty Proteasomes That Are Inhibited by Ecm29 Protein 2011)
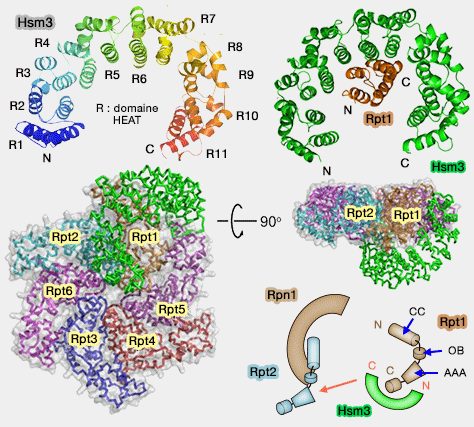
- Hsm3, Rpt1, par 11 répétitions HEAT (Structural Basis for Specific Recognition of Rpt1p, an ATPase Subunit of 26 S Proteasome, by Proteasome-dedicated Chaperone Hsm3p 2012). Il contacte aussi la domaine AAA de Rpt2, Rpt2 étant lié lui-même par son domaine N-terminal avec Rpn1, d'où le complexe Hsm3/Rpt1/Rpt2/Rpn1 (Dual functions of the Hsm3 protein in chaperoning and scaffolding regulatory particle subunits during the proteasome assembly 2012).
(Figure : vetopsy.fr d'après Kim et Nakamura)
Comme pour les protéines chaperons du coeur catalytique, ces protéines ont pour rôle de prévenir un assemblage précoce avec le coeur catalytique par leur fixation sur les extrémités C-terminales des Rpt
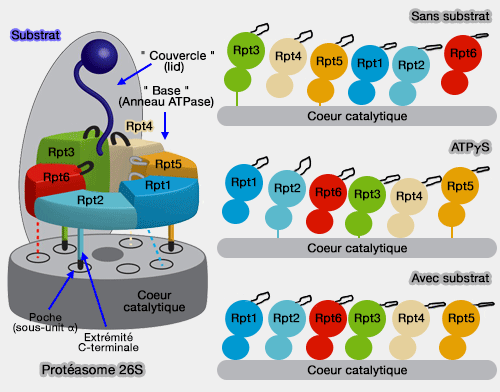
- Pour ce qui est de Rpt3 (Nas6) et de Rpt5 (Nas2), il semble que ce soit le motif HbYX qui soit reconnu pour prévenir sa liaison avec les poches du coeur catalytique entre les sous-unités α - cf. positionnement de l'anneau ATPase - (Chaperone-mediated pathway of proteasome regulatory particle assembly 2009).
- Cet assemblage de dimères est conforme à ce que l'on observe dans l'hexamère mature, par exemple au niveau des 3 superhélices (coiled coil) formées par les Rpt (Hexameric assembly of the proteasomal ATPases is templated through their C termini 2009).
Ordre d'assemblage
1. Les complexes Rpt4/Rpt5/Nas2 et Rpt3/Nas6/Rpt6/Rpn14 s'associent en premier.

(Figure : vetopsy.fr d'après Tomko)
Cet ordre d'incorporation paraît le bon, du moins chez la levure (Heterohexameric Ring Arrangement of the Eukaryotic Proteasomal ATPases: Implications for Proteasome Structure and Assembly 2010).
Chez les Mammifères, il semble qu'on retrouve des complexes sans Nas2 (p27) avec Rpt4 et Rpt5 (Assembly pathway of the mammalian proteasome base subcomplex is mediated by multiple specific chaperones 2009 et Multiple proteasome-interacting proteins assist the assembly of the yeast 19S regulatory particle 2009.
2. Puis, ce complexe agrège le module Rpt1/Rpt2/Rpn1/Hsm3, en éliminant Nas2.
3. Quand le couvercle se fixe sur la base par Rpn12 (cf. plus bas) ou lors de la fixation de 19S sur le coeur catalytique 20S selon les auteurs, les autres chaperons (Hsm3, Nas6 et Rpn14) se dissocient.
(Figure : vetopsy.fr d'après Estin et Bhattacharyya)
Deux autres molécules peuvent intervenir pour faciliter l'assemblage.
- Ubp6, une désubiquitinase chez la levure, s'associe avec le complexe Hsm3 pour désubiquitiner les protéines de ce module (The Catalytic Activity of Ubp6 Enhances Maturation of the Proteasomal Regulatory Particle 2011).
- TNSK (tankyrase (ADP-ribosyltransférase) régule l'activité du protéasome en séquestrant p27 et S5b et en favorisant l'assemblage de la base (Proteasome Regulation by ADP-Ribosylation 2013).
Action du coeur catalytique
Il semble que le coeur catalytique puisse servir de protéine chaperon ou d'échafaudage (scaffold protein) pour la formation de la base (Molecular Architecture and Assembly of the Eukaryotic Proteasome 2013).
- D'une part, les Rpt se fixent dans des poches entre les sous-unités α (cf. positionnement de l'anneau ATPase) et pourraient servir de modèle pour les assembler correctement, en particulier Rpt6.
-
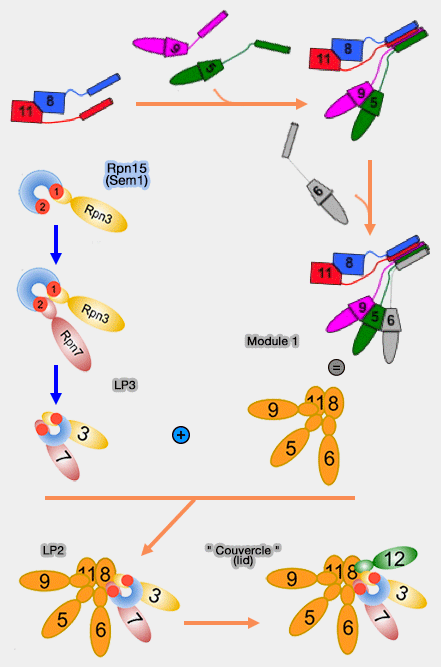
Assemblage du " couvercle " (lid)
(Figure : vetopsy.fr d'après Estrin et Tomko)
Assemblage du
" couvercle "
Rpn15, appelé aussi DSS1 chez les Mammifères et Sem1 chez la levure, participe à l'assemblage de l'ensemble du " couvercle " (lid), mais n'est pas indispensable dans le protéasome mature (The Intrinsically Disordered Sem1 Protein Functions as a Molecular Tether During Proteasome Lid Biogenesis 2014).
1. Le module 1 est formé par Rpn5, 6, 8, 9 et 11 : les étapes d'incorporation des différentes sous-unités ne sont pas claires (Dissection of the assembly pathway of the proteasome lid in Saccharomyces cerevisiae 2010). Il semblerait que Rpn6 se lie en dernier.
2. Sem1 se lie d'abord à Rpn3, puis à Rpn7, par les sites acides situés de chaque côté de cette protéine : ce module 2 prend une forme de fer à cheval et est nommé LP3 (Lid Particle 3).
3. LP3 se lie au module 1 pour former LP2.
4. LP2 peut alors se lier à Rpn12 pour former le " couvercle " (lid).
Rpn12, par son extrémité C-terminale, permet la liaison du " couvercle " à la base (Incorporation of the Rpn12 subunit couples completion of proteasome regulatory particle lid assembly to lid-base joining 2011).
-
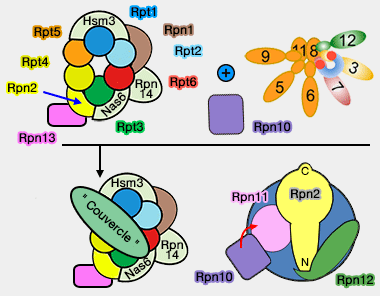
Assemblage du " couvercle " (lid) et de la base
(Figure : vetopsy.fr d'après Estrin et Tomko) - Cette liaison est accompagnée par celle de Rpn10, situé à l'interface entre la base et le " couvercle ".
- Rpn12 et Rpn10 semble être redondants pour stabiliser la liaison entre la base et le " couvercle ". Ils se placent de chaque côté de Rpn2. Ils pourraient agir via la liaison de Rpn12 avec Rpn2, et celle de Rpn10 qui interagit avec Rpn11, lui-même lié à Rpn2.
Toutefois, un complexe comprenant un couvercle complet lié à Rpt3, Rpt6, Rpn2, et la désubiquitinase Uch37 a été observé (Subcomplexes of PA700, the 19 S regulator of the 26 S proteasome, reveal relative roles of AAA subunits in 26 S proteasome assembly and activation and ATPase activity 2009).
Pour l'instant, le processus intime est encore largement inconnu.
- Il semblerait que les hélices C-terminales des Rpn ordonnent l'ordre d'incorporation des différentes sous-unités (Formation of an Intricate Helical Bundle Dictates the Assembly of the 26S Proteasome Lid 2013).
- D'autres protéines chaperons, qui ne sont pas toutes répertoriées, semblent intervenir dans ce processus comme Hsp90 (Complete subunit architecture of the proteasome regulatory particle 2012) ou Yin6/INT6.
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


