Constituants cellulaires : protéasome 26S
Structure du complexe 19S : " couvercle " (lid)
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Le couvercle " (lid) qui, avec la base compose le complexe régulateur 19S (RP : Regulatory Particle)attache lui aussi le complexe 19S au coeur catalytique 20S (CP : Core Particle).
Vue d'ensemble du couvercle
Le couvercle conditionne l’entrée dans la chambre catalytique en modulant l'activité enzymatique des différentes sous-unités du protéasome (Complete subunit architecture of the proteasome regulatory particle 2012).
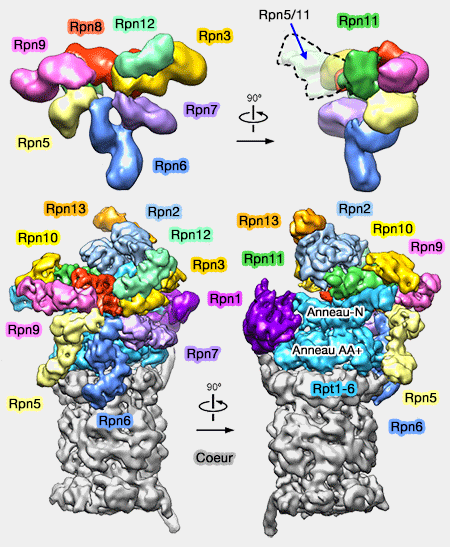
(Figure : vetopsy.fr d'après Lander)
1. Le " couvercle " est composé de 8 protéines qui possèdent toutes des hélices C-terminales formant un domaine coiled-coil (superhélice) qui les connectent toutes ensemble (Formation of an Intricate Helical Bundle Dictates the Assembly of the 26S Proteasome Lid 2013).
Chacune de ces huit sous-unités a des paralogues dans des complexes qui adoptent une architecture similaire (Architecture of Human Translation Initiation Factor 3 2013).
- COP9/signalosome (CSN) - The COP9 signalosome: more than a protease 2008 et Crystal structure of the human COP9 signalosome 2014 - inactive les E3 ubiquitine ligases Cullin-RING (CRL) en les séparant de leur activateur NEDD8, fixé de manière covalente à la culline.
- Le facteur d'allongement d'initiation 3 (eIF3) a un rôle essentiel dans la traduction, processus par lequel l'ARN messager est traduit en protéines chez les eucaryotes (initiation, élongation, terminaison et recyclage)
2. L'assemblage du complexe régulateur est favorisé par des protéines chaperons et s'effectue indépendamment pour la base et le " couvercle ".
Rpn à domaine PCI : Rpn3, 5, 6, 7, 9, 12
6 sous-unités - Rpn3, 5, 6, 7, 9, 12 - possèdent :
- un domaine PCI (Proteasom, COP9/signalosome, eIF3 ou Cyclosome-Initiation),
- une longue hélice C-terminale (Unveiling the Long-Held Secrets of the 26S Proteasome 2013).
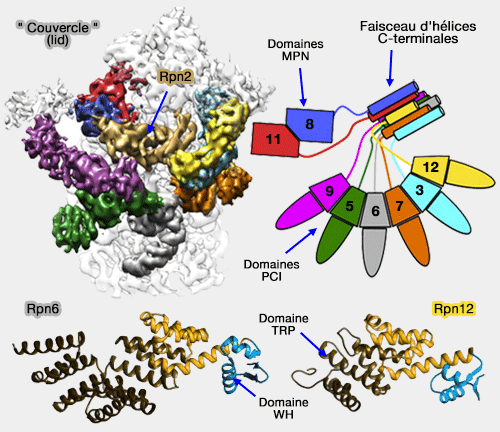
(Figure : vetopsy.fr d'après Unverboden et Estrin et Förster)
1. Leurs domaines PCI forment une sorte de " fer à cheval ".
- Leurs hélices C-terminales forment un paquet de superhélices qui est lié à Rpn2 et à Rpn11/8.
- Rpn11 et Rpn8, son partenaire, sont au milieu des branches du fer à cheval et proches de Rpn2.
2. Rpn5, Rpn6 et Rpn7 s'étendent sur le coeur catalytique et le contactent directement.
- Rpn5 et Rpn6, par leur répétition tétratricopeptide (TPR : tetratricopeptide repeat) N-terminale, interagissent, respectivement, avec Rpt6/3 de la base et les sous-unités α1 et α2 du coeur catalytique.
-
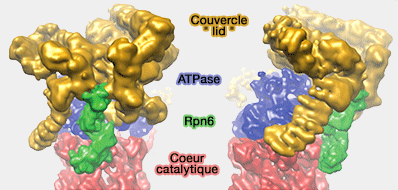
Rpn6 et association couvercle/coeur
(Figure : vetopsy.fr d'après Pathare)

Rpn6 semble jouer un rôle majeur dans la stabilisation du complexe 19S/20S(The proteasomal subunit Rpn6 is a molecular clamp holding the core and regulatory subcomplexes together 2011).
Dimère Rpn11/8 à domaine MPN
Deux sous-unités - Rpn11 et 8 - comprennent un domaine MPN - Mpr1/Pad1 N-terminal - (Structure of the Jab1/MPN Domain and Its Implications for Proteasome Function 2003 et Prediction of a common structural scaffold for proteasome lid, COP9-signalosome and eIF3 complexes 2005 et Crystal structure of the proteasomal deubiquitylation module Rpn8-Rpn11 2014)
1. Rpn11 est une désubiquitinase (DUB) qui possède une activité métalloprotéase MPN/JAMM.
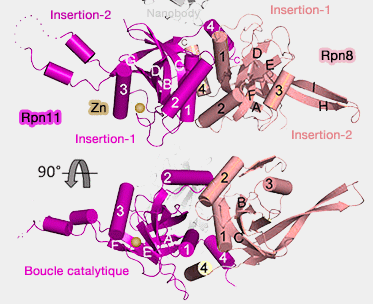
(Figure : vetopsy.fr d'après Pathare)
Cette famille contient un glutamate conservé en amont d'une boucle comprenant le zinc catalytique, H(S/T) HX7SXXD (cf mécanisme de protéolyse des métalloprotéases), révélé dans la structure de AfJAMM, un homologue chez les Archées (JAMM: A Metalloprotease-Like Zinc Site in the Proteasome and Signalosome 2004 et Structural basis for specific cleavage of Lys 63-linked polyubiquitin chains 2008).
- Rpn11 est situé sur le côté l'anneau OB de la base, au-dessus de la superhélice des Rpt5/4 et son site catalytique est inactif.
- Rpn11 contacte une grande surface de Rpn2, en particulier dans sa portion centrale, et le faisceau d'hélices C-terminales des autres Rpn du couvercle.
- Le dépliage et la translocation du substrat vers le coeur catalytique sont couplées à la désubiquitination.
Rpn11 est inactif, comme le " couvercle " par ailleurs, s'il est isolé : il s'active par le cycle ATPasique de la base (cf. information supplémentaire).
Des désubiquitinases supplémentaires peuvent s'associer de manière réversible au protéasome.
2. Rpn8, son partenaire de liaison, n'a pas d'activité catalytique, ne possédant pas le module MPN/JAMM.
Rpn15, appelé aussi DSS1 chez les Mammifères et Sem1 chez la levure, considérée souvent comme la neuvième sous-unité, participe à l'assemblage du " couvercle ", mais n'est pas indispensable dans le protéasome mature.
Protéines associées au complexe 19S
De nombreuses protéines sont associées au complexe 19S, en se liant le plus souvent à Rpn1, pour moduler la dégradation du substrat.
Les principales sont :
- Rad23, DSK2 et Ddi1, récepteurs alternatifs des ubiquitines ;
- Ubp6 (levure), Uch37 et USP14 (Mammifères), désubiquitinases ;
- Hul5/KIAA10 (E3 ubiquitine ligase HECT) dont le rôle paraît complexe.
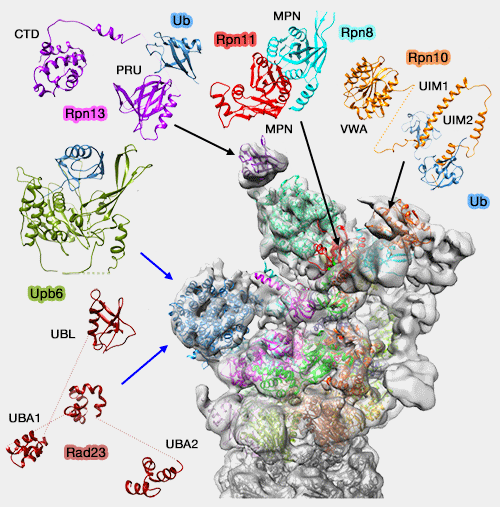
(Figure : vetopsy.fr d'après Kish-Trier)
Assemblage du protéasome
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosquelettemicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


