Domaines protéiques
Motifs à doigt de zinc (zinc-finger)
: vue d'ensemble

Les motifs à doigt de zinc sont de petits motifs structuraux d'une trentaine d'acides aminés trouvés dans les protéines et dont le rôle est de stabiliser leurs plis par un ou plusieurs ions Zn++.
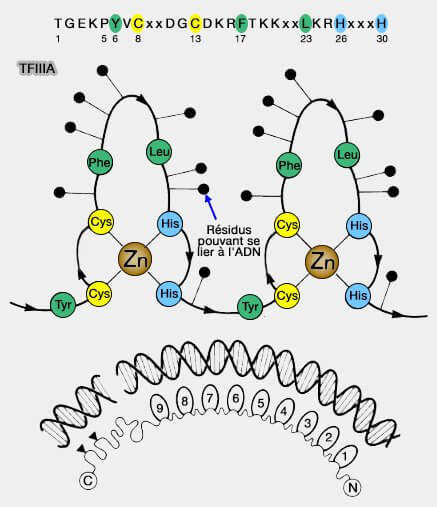
(Figure : vetopsy.fr d'après Miller)
Ils ont été identifiés pour la première fois dans TFIIIA, un facteur de transcription (TF avec le 5S ARN) du xénope qui comporte neuf séquences en tandem de 30 acides aminés qui permettent la formation de boucles se liant à l'ADN qui ressemblent à des doigts, d'où leur nom (Repetitive zinc-binding domains in the protein transcription factor LiA from Xenopus oocytes 1985 et RNA and DNA binding zinc fingers in Xenopus TFIIIA 1992).
Vue d'ensemble
1. Le motif à doigt de zinc classique ancestral, présent dans le Last Universal Common Ancestor of Life (LUCA), est un domaine treble-clef (" clef de sol ") qui ne comporte qu'un seul ion zinc chélaté par 4 cystéines - (Functional diversification of the RING finger and other binuclear treble clef domains in prokaryotes and the early evolution of the ubiquitin system 2011).
Deux ligands forment une articulation et deux autres forment l'extrémité N-terminale d'une hélice.
Les cystéines et le métal chélaté peuvent être perdus comme dans les U-box, ou certains types de nucléases comme les HNH/EndoVII-fold nucléases (A novel immunity system for bacterial nucleic acid degrading toxins and its recruitment in various eukaryotic and DNA viral systems 2011).
2. Ce domaine treble-clef peut rester :
a. mononucléaire, c'est-à-dire ne garder qu'un seul site de chélation du zinc,
b. binucléaire, en acquérant un deuxième site de chélation du zinc qui est retrouvé dans de nombreux domaines (Treble clef finger—a functionally diverse zinc-binding structural motif 2001) :
-
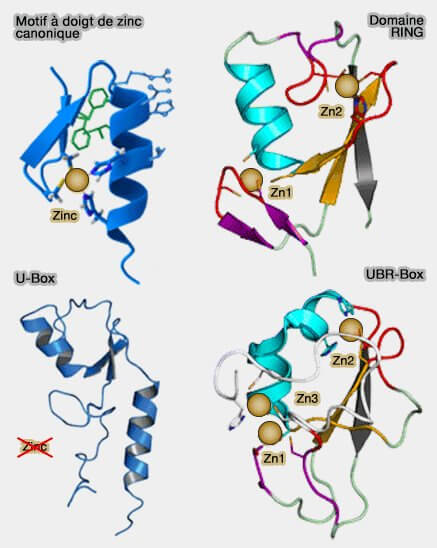
Différents domaines à motifs à doigt de zinc
(Figure : vetopsy.fr) - les domaines LIM,
- les domaines PHD,
- les B-box,
- les domaines UBP (The Ubiquitin Binding Domain ZnF UBP Recognizes the C-Terminal Diglycine Motif of Unanchored Ubiquitin 2006),
- les domaines FYVE et ZZ,
- les domaines ester/diaglycerol-binding C1 (Crystal structure of the Cys2 activator-binding domain of protein kinase Cδ in complex with phorbol ester 1995),
Remarque : on retrouve le domaine treble-clef d'autres domaines comme les domaines :
- AN1 (Two related localized mRNAs from Xenopus laevis encode ubiquitin-like fusion proteins 1993 et (Genome-wide analysis of the stress associated protein (SAP) gene family containing A20/AN1 zinc-finger(s) in rice and their phylogenetic relationship with Arabidopsis 2006),
- MYND - MYeloid, Nervy, and DEAF-1 - (Structural Basis for Recognition of SMRT/N-CoR by the MYND Domain and Its Contribution to AML1/ETO's Activity 2007),
- UPF1 (Crystal structure of the UPF2-interacting domain of nonsense-mediated mRNA decay factor UPF1 2006).
Structures et fonctions des motifs à doigts dde zinc
Biologie cellulaire et moléculaireConstituants de la celluleMatrice extracellulaireReproduction cellulaireBiochimieChimie organiqueBioénergétiqueProtidesAcides aminésProtéinesDomaines protéiquesModifications post-traductionnelles des protéinesDégradations des protéinesUbiquitinationGlucidesLipidesEnzymesCoenzymesVitaminesHormonesTransport membranaireMoteurs moléculairesVoies de signalisation


