Biologie cellulaire : peroxysomes
Biogenèse et pexophagie
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Morphoplasme : organites
- Système endomembranire
- Mitochondries
- Peroxysomes
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

Les peroxysomes sont de petits organites limités par une membrane qui se trouvent à l’intérieur des cellules.
- Les peroxysomes participent à la respiration cellulaire, comme les mitochondries, i.e. ils consomment de l'oxygène en employant des enzymes oxydatives.
- Leur nom provient de la leur capacité à neutraliser le peroxyde d'hydrogène (H202) très toxique, pour produire de l’eau.
C-Biogenèse des peroxysomes

Vous pouvez lire : Comparative Genomics of Peroxisome Biogenesis Proteins: Making Sense of the PEX Proteins (2021) et Peroxisome biogenesis, membrane contact sites, and quality control (2022).
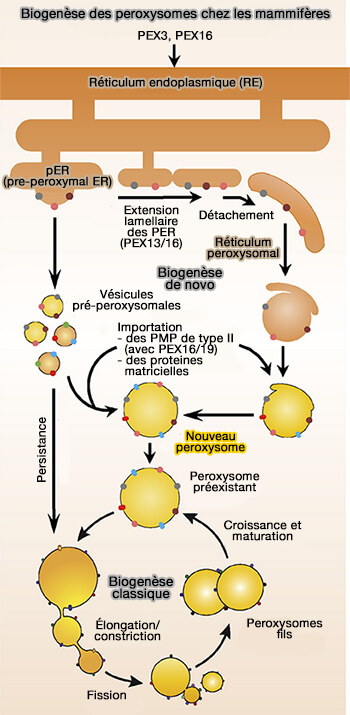
La formation des peroxysomes peut emprunter deux voies :
(Figure : vetopsy.fr d'après Agrawal et coll)
- la voie alternative de formation de novo,
- la voie classique de croissance et de division des organites préexistants, voie prédominante.
Voie alternative : formation de novo
Dans la voie alternative des cellules de mammifères, le réticulum endoplasmique (RE) et les mitochondries peuvent contribuer à la formation de novo de peroxysomes (De novo peroxisome biogenesis: Evolving concepts and conundrums 2016).

Le ciblage initial des protéines membranaires peroxisomales (PMP) en l'absence de peroxysomes semble être un événement clé dans la formation de novo (Unloosing the Gordian knot of peroxisome formation 2018).
1. Plusieurs PMP clés (par exemple Pex16, Pex3) ciblent le réticulum endoplasmique (RE)où elles sont séquestrées dans des vésicules pré-peroxysomales qui sont libérées pour formenr des peroxysomes qui se développent et se divisent ensuite pour se multiplier.
- Des protéines de type réticulon Pex30 et Pex31 sont impliquées dans la génération d'un sous-domaine du RE dans lequel bourgeonnent les vésicules pré-peroxysomales (A family of membrane-shaping proteins at ER subdomains regulates pre-peroxisomal vesicle biogenesis 2016).
- Les protéines de l'ESCRT-III, Vps20 (CHMP6) et Snf7 (CHMP4A/B/C) joueraient un rôle dans la libération de vésicules pré-peroxysomales du RE, soutenant l'origine des vésicules pré-peroxysomales (ESCRT-III is required for scissioning new peroxisomes from the endoplasmic reticulum 2018).
En outre, les peroxysomes sont des organites semi-autonomes qui dépendent d'autres compartiments cellulaires comme le RE pour obtenir des lipides ou encore des protéines spécifiques (Origin and spatiotemporal dynamics of the peroxisomal endomembrane system 2014).
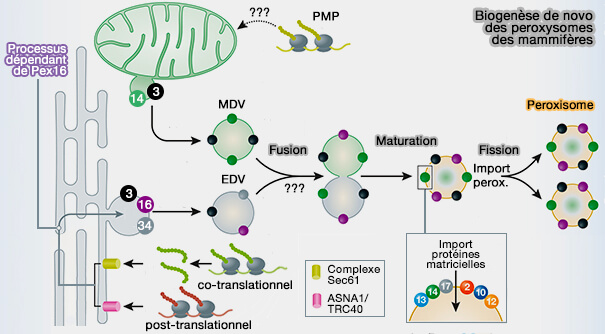
2. Dans les fibroblastes humains dépourvus de Pex3 ou Pex16 dépourvus de peroxysomes, leur réintroduction a engendré deux processus (Newly born peroxisomes are a hybrid of mitochondrial and ER-derived pre-peroxisomes 2017) :
- L'introduction de Pex3 a ciblé les mitochondries pour former des vésicules pré-peroxysomales ou MDV (Mitochondria-Derived Vesicles).
- L'introduction de Pex16 a ciblé le réticulum endoplasmique (RE) pour former des vésicules pré-peroxysomales ou EDV (ER-derived vesicles).
- Les EDV et les MDV fusionnent pour générer de nouveaux peroxysomes capables d'importer des proteines peroxysomales.
En présence de peroxysomes, les vésicules pré-peroxysomales peuvent fusionner avec des peroxysomes en croissance ou existants pour fournir certaines protéines et certains lipides.
(Figure : vetopsy.fr d'après Farré et coll)
Voie classique
Dans la voie classique, les peroxysomes grossissent et subissent une fission pour former deux nouveaux peroxysomes.
1. Les peroxysomes forment un réseau dynamique et peuvent se multiplier par croissance membranaire et division d'organites préexistants (Proliferation and fission of peroxisomes - An update 2016).
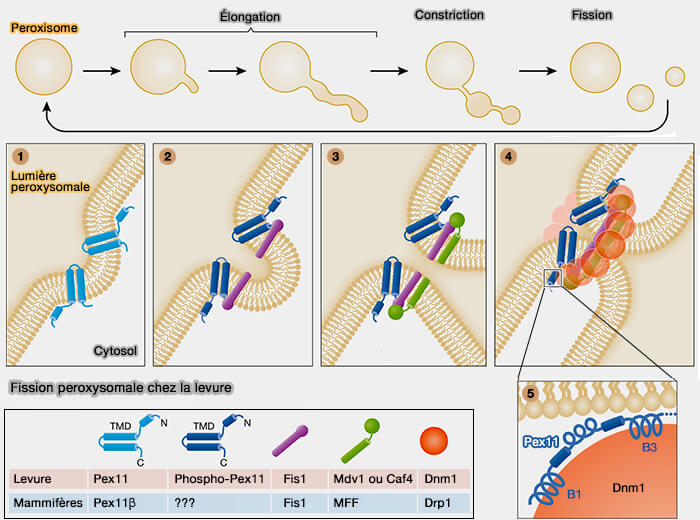
Cette voie classique implique (
- le remodelage et l'expansion de la membrane peroxysomale par la formation d'extensions de membrane tubulaire,
- leur constriction,
- la fission en deux nouveaux compartiments membranaires,
- l'importation ultérieure de protéines matricielles nouvellement synthétisées (Peroxisome Dynamics in Cultured Mammalian Cells 2009).

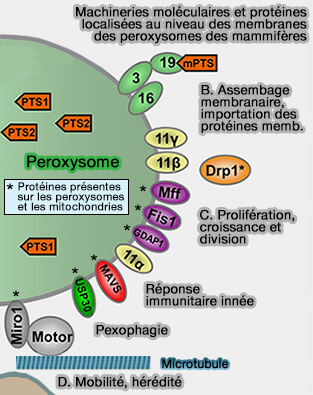
Pex11α, Pex11β et Pex11γ sont impliquées dans la régulation de la taille et du nombre de peroxysomes.
2. La peroxine membranaire Pex11β est un facteur clé dans la régulation du nombre de peroxysomes chez les mammifères, qui est désormais associée à toutes les étapes de la croissance et de la division des peroxysomes (Pex11pβ-mediated growth and division of mammalian peroxisomes follows a maturation pathway 2010).
La famille Pex11 comprend trois membres Pex11α, β et γ (PEX11 family members are membrane elongation factors that coordinate peroxisome proliferation and maintenance 2010).
(Figure : vetopsy.fr d'après Farré et coll)
Pex11β, par son oligomérisation et son interaction avec les lipides membranaires via des hélices amphipathiques N-terminales, remodèle, déforme et allonge la membrane peroxysomale avant la fission (Pex11mediates peroxisomal proliferation by promoting deformation of the lipid membrane 2015 et The N-terminal amphipathic helix of Pex11p self-interacts to induce membrane remodelling during peroxisome fission 2018).
Pex11 serait recruté par Pex19 (Recognition and Chaperoning by Pex19, Followed by Trafficking and Membrane Insertion of the Peroxisome Proliferation Protein, Pex11 2021)
(Figure : vetopsy.fr d'après Islinger et coll)
Pex11β, par sa phosphorylation sur S173, interagit avec Fis1, la protéine de fission mitochondriale (Fission protein1) et Mff (Mitochondrial Fission Factor) au niveau de la membrane peroxysomale (
- Ces adaptateurs recrutent alors la dynamine GTPase Drp1, contribuant ainsi à l'assemblage de la machinerie de division peroxysomale (Mff functions with Pex11pβ and DLP1 in peroxisomal fission 2013).
- En outre, Pex11β fonctionne comme une GAP (GTPase-Activating Protein ou GTPase-Accelerating Protein) pour Drp1 lors de la fission peroxysomale (The membrane remodeling protein Pex11p activates the GTPase Dnm1p during peroxisomal fission 2015).
Chez la levure, Fis1 recrute les récepteurs périphériques, Mdv1 et/ou Caf4 qui assemblent un anneau de Dnm1 autour du site de constriction des peroxysomes. Dmn1 interagit avec Fis1 et deux hélices de Pex11 nommées B1 et B3. L'hydrolyse du GTP par Dnm1, renforcée par l'interaction avec l'hélice B3 de Pex11, conduit à une constriction qui divise le peroxysome.
Pex11β pourrait être également impliqué dans la constriction.
3. Les peroxysomes et les mitochondries partagent ainsi des protéines de leur machinerie de division, i.e. Fis1, Mff, Drp1 et GDAP1 - Ganglioside-induced Differentiation-Associated Protein 1 - (Charcot-Marie-Tooth disease-associated mutants of GDAP1 dissociate its roles in peroxisomal and mitochondrial fission 2013 et Systematic mapping of contact sites reveals tethers and a function for the peroxisome-mitochondria contact 2018)
Pour les mitochondries, le rôle du RE dans la division membranaire a été mis en évidence (
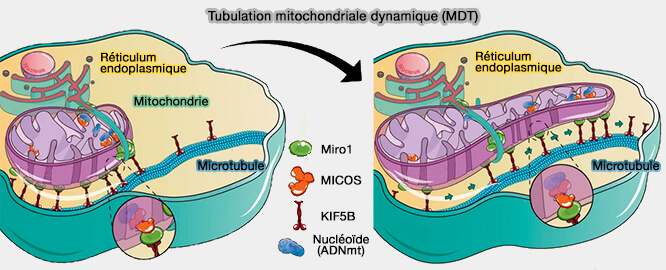
(Figure : vetopsy.fr d'après Elbaz-Alon et coll)
Le partage des composants de division entre les peroxysomes et les mitochondries est considéré comme une stratégie commune conservée au cours de l'évolution chez les mammifères, les champignons et les plantes, contribuant à la " connexion peroxysome-mitochondrie ", qui a un impact sur leurs fonctions coopératives et leur contribution aux maladies (The Peroxisome-Mitochondria Connection: How and Why? 2017).
Certaines maladies sont dues à des défauts de la division peroxysomale impliquant Drp1, Mff et Pex11β (
Pexophagie
La dégradation sélective des peroxysomes ou pexophagie s'effectue par autophagie.

La pexophagie est bien décrite dans : Pexophagy in yeast and mammals: an update on mysteries (2018).
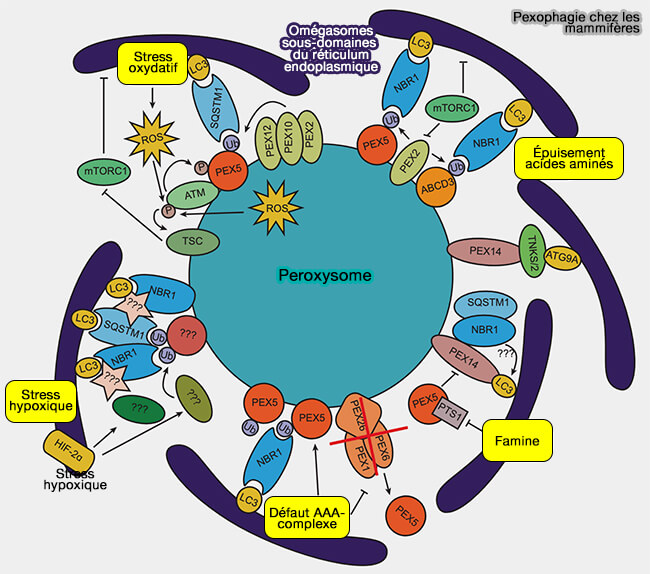
(Figure : vetopsy.fr d'après Eberhart et Kovacs)
La formation de l'autophagosome est régulée par UNC-51-like kinase 1/2 (ULK1/2, les orthologues d'ATG1 de la levure) et par les complexes de classe III phosphatidylinositol 3-kinase (PI3K).
- Le complexe Ulk se compose d'ULK1/2, ATG13, FIP200 (ATG17), ATG101 et subit une translocation lors de l'activation de sous-domaines du réticulum endoplasmique (RE) pour former des vésicules pré-peroxysomales ou EDV (ER-derived vesicles), appelé omégasomes, pour conduire la nucléation des autophagosomes (Structure and Function of the ULK1 Complex in Autophagy 2016).
- Le complexe Ulk active le complexe PI3KC3-CI, qui se compose de VPS34, Beclin1 (ATG6 ou Vps30), ATG14, et Vps15/p150, conduisant à sa translocation au RE, où il produit du PI(3,4,5)P3 ou PIP3 pour conduire la formation d'omégasomes.
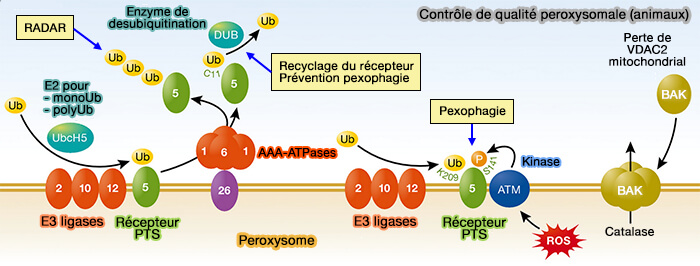
(Figure : vetopsy.fr d'après Farré et coll)
Motilité et MCS des peroxysomes des peroxysomes
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeMitochondriesSystème endomembranaireRéticulum endoplasmiqueAppareil de GolgiEndosomesLysosomesPeroxysomesProtéasomesCytosqueletteMicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaireMoteurs moléculairesVoies de signalisation


