Constituants cellulaires : protéasome 26S
Base du complexe régulateur 19S : les Rpn
- Biologie cellulaire et moléculaire
- Constituants de la cellule
- Membrane plasmique
- Noyau
- Cytoplasme
- Hyaloplasme
- Cytosol
- Cytosquelette
- Proteasome
- Vue d'ensemble
- Structure générale du protéasome 26S
- Assemblage du protéasome 26S
- Fonctionnement du protéasome
- Morphoplasme : organites
- Hyaloplasme
- Matrice extracellulaire
- Reproduction cellulaire
- Biochimie
- Transport membranaire
- Moteurs moléculaires
- Voies de signalisation

La base, qui, avec le " couvercle " (lid), compose le complexe régulateur 19S (RP : Regulatory Particle-), comprend 10 protéines.
La base, en contact avec le coeur catalytique 20S (CP : Core Particle) comprend (Base-CP proteasome can serve as a platform for stepwise lid formation 2015) :
- 6 Rpt : 1 à 6 (Regulatory particle triple-A protein ou Regulatory particle triphosphatase)
- 4 Rpn : 1, 2, 10, 13 (Regulatory particle non-ATPase).
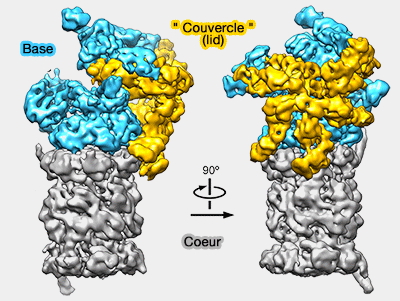
(Figure : vetopsy.fr d'après Lander)
Le protéasome est une structure dynamique qui modifie sa conformation durant le processus et qui interagit avec un grand nombre de protéines.
- La description ci-dessous concerne surtout la nomenclature, la structure, la conformation des différentes protéines : les positions décrites sont celles supposées du protéasome libre de tout substrat.
- Les changements conformationnels ou positionnels sont décrits dans le fonctionnement du protéasome.
L'assemblage du complexe régulateur est favorisé par des protéines chaperons et s'effectue indépendamment pour la base et le " couvercle ".
Rpn1, 2, 10, 13 de la base du 19S
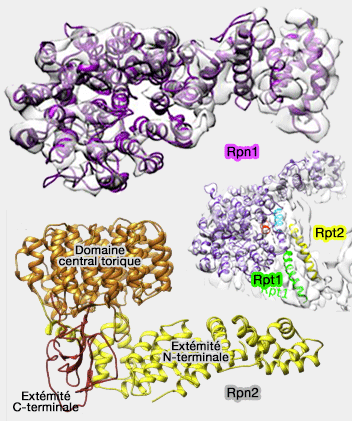
(Figure : vetopsy.fr d'après Unverboden et Föerster)
Les protéines de la base, autres que les Rpt, ainsi que celles du " couvercle " (lid), sont appelées Rpn - Regulatory particle non-ATPase - (Near-atomic resolution structural model of the yeast 26S proteasome 2012).
Rpn1 et Rpn2
Rpn1 et Rpn2 , protéines d'échafaudage (scaffold protein), sont relativement similaires (40% d'homologie).
- Elles forment chacune un solénoïde grâce à leurs répétitions tétratricopeptide (TPR : tetratricopeptide repeat) et possèdent deux sous-unités liées par un lien flexible (What Curves α-Solenoids? Evidence for an α-HELICAL toroid structure of Rpn1 and Rpn2 proteins of 26 S proteasome 2002).
- Elles diffèrent par leur extrémité N-terminale qui est en rotation de 40° autour d'un lien flexible - 153-175 et 95-119 respectivement - (Electron microscopic evidence in support of alpha-solenoid models of proteasomal subunits, Rpn1 and Rpn2 2009).
Rpn1 est la plus grande des sous-unités du protéasome.
Rpn1 et Rpn2 contactent l'anneau OB (ou N).
- Rpn1 le fait trois hélices : 2 pour respectivement pour la superhélice Rpt1/2, la troisième entrant vraisemblablement dans la composition d'une superhélice (Deep classification of a large cryo-EM dataset defines the conformational landscape of the 26S proteasome 2014).
-
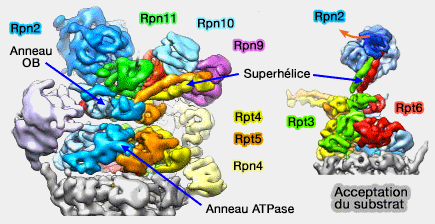
Liaisons entre les sous-unités du complexe régulateur
(Figure : vetopsy.fr d'après Bhattacharyya)
Rpn1 et Rpn2 se lient aussi avec des protéines liées à l'ubiquitine,
- Rpn10 et Rpn13, mais aussi des protéines associées au protéasome pour Rpn1,
- Rpn13 pour Rpn2 (Rpn1 and Rpn2 Coordinate Ubiquitin Processing Factors at Proteasome 2012).
Rpn10 et Rpn13
Rpn10 et Rpn13 sont des ligands de l'ubiquitine, placés à la périphérie et proches de la région distale, position appropriée pour capturer les substrats (Near-atomic resolution structural model of the yeast 26S proteasome 2012 et Localization of the proteasomal ubiquitin receptors Rpn10 and Rpn13 by electron cryomicroscopy 2012 et Together, Rpn10 and Dsk2 can serve as a polyubiquitin chain-length sensor 2009)
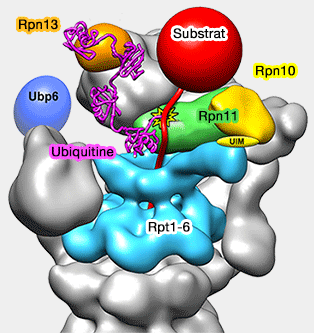
du substrat et sa désubiquitination
(Figure : vetopsy.fr d'après Lander)
1. Rpn10 contient un (levure) ou deux (Mammifères) domaines UIM, domaine UBD de liaison à l'ubiquitine et un domaine VWA (Von Willebrand factor type A).
- Rpn10 se trouve à l'interface couvercle/base.
- Il est au contact de Rpn11 et Rpn9 et stabilise Rpn11, la désubiquitinase (DUB) de la famille des MPN/JAMM, lié à Rpn2 qui stabilise sa position au milieu du solénoïde de Rpn1.
- Le domaine UIM de Rpn10 contacte la superhélice de Rpt4/5, en stabilisant sa position par rapport aux autres sous-unités.
Pour certains, Rpn10 ne fait pas partie ni de la base, ni du " couvercle " et se lie uniquement au protéasome 26S lors de son assemblage (Crystal structure of the proteasomal deubiquitylation module Rpn8-Rpn11 2014).
2. Rpn13 :
- contient un domaine PRU, domaine UBD de liaison à l'ubiquitine.
- Rpn13 est lié directement à Rpn2 et est situé le plus loin possible du centre du protéasome.
" Couvercle " (lid)
Biologie cellulaire et moléculaireMembrane plasmiqueNoyauCytoplasmeRéticulum endoplasmiqueAppareil de GolgiMitochondriesEndosomesLysosomesPeroxysomesProtéasomesCytosqueletteMicrofilaments d'actineFilaments intermédiairesMicrotubulesMatrice extracellulaireReproduction cellulaireBiochimieTransport membranaire Moteurs moléculairesVoies de signalisation


